 |
 |

PAGES DU PRATICIEN
Association entre les génotypes de la CETP (Cholesteryl Ester Transfer Protein) et la masse et l’activité de la CETP, les taux de lipides et le risque coronaire
Alexander Thompson, MRes, MPhil;
Emanuele Di Angelantonio, MD, MSc;
Nadeem Sarwar, MRPharmS, MPhil;
Sebhat Erqou, MD, MPhil;
Danish Saleheen, MBBS, MPhil;
Robin P. F. Dullaart, MD, PhD;
Bernard Keavney, MD, FRCP;
Zheng Ye, PhD;
John Danesh, DPhil, FRCP
JAMA. 2008;299(23):2777-2788
RÉSUMÉ
 |  |
Contexte L'importance de la voie de la CETP (cholesteryl ester transfer protein) dans la maladie coronarienne est incertaine. L'étude des génotypes de la CETP peut aider à mieux comprendre la pertinence de cette voie par rapport au métabolisme des lipides et au risque de maladie coronarienne.
Objectif Evaluer l’association des génotypes de la CETP avec les phénotypes de la CETP, les taux de lipides, et le risque coronaire.
Sources des données Etudes publiées entre janvier 1970 et janvier 2008, identifiées par des recherches sur ordinateur et manuelles dans MEDLINE, EMBASE, BIOS, Science Citation Index, et dans la Chinese National Knowledge Infrastructure Database. Les études précédemment non publiées ont été cherchées en correspondant avec les investigateurs.
Sélection des études Études pertinentes liées principalement à trois polymorphismes communs de la CETP (TaqIB [rs708272], I405V [rs5882], et –629C>A [rs1800775]) et à 3 polymorphismes rares de la CETP (D442G [rs2303790], –631C>A [rs1800776], et R451Q [rs1800777]).
Recueil des données Les informations sur les génotypes de la CETP, les phénotypes de la CETP, les taux de lipides, la maladie coronarienne, et les caractéristiques des études ont été recueillies dans les publications, fournies par les investigateurs, ou les deux.
Résultats Quatre-vingt-douze études avaient des données sur les phénotypes de la CETP, les taux de lipides, ou les deux pour 113 833 participants en bonne santé, et 46 études avaient des données sur 27 196 cas coronaires et 55 338 témoins. Pour chaque allèle A transmis, les personnes ayant le polymorphisme de TaqIB avaient une masse moyenne inférieure de CETP (–9.7% ; intervalle de confiance à 95% [IC], –11.7% à –7.8%), une diminution de l'activité moyenne de la CETP (–8.6% ; IC 95%, –13.0% à –4.1%), des concentrations moyennes plus élevées du cholestérol des lipoprotéines de haute densité (HDL-C) (4.5% ; IC 95%, 3.8%-5.2%), et des concentrations moyennes plus élevées de l'apolipoprotéine AI (2.4% ; IC 95%, 1.6%-3.2%). Le profil des résultats a été très similaire pour les polymorphismes I405V et –629C>A. Les rapports combinés de cotes par allèle (OR) de maladie coronarienne étaient de 0.95 (IC 95%, 0.92-0.99) pour TaqIB, de 0.94 (IC 95%, 0.89-1.00) pour I405V, et de 0.95 (IC 95%, 0.91 - 1.00) pour –629C>A.
Conclusions Trois génotypes de la CETP associés à une inhibition modérée de l’activité de la CETP (et, en conséquence, à des taux légèrement plus élevés de HDL-C) montrent une faible inversion de des associations avec le risque coronaire. Les OR de maladie coronarienne étaient compatibles avec les réductions attendues du risque que l’on observe avec des augmentations équivalentes de concentration du HDL-C dans les études prospectives disponibles.
Les études d'observation et expérimentales menées chez l'homme et l’animal ont encouragé le développement d’agents pharmacologiques qui augmentent les taux de cholestérol des lipoprotéines de haute densité (HDL-C) dans la prévention de la maladie coronarienne.1-8 Ces agents incluent les inhibiteurs de la CETP (cholesteryl ester transfer protein), une protéine qui facilite l'échange des cholestéryl esters des triglycérides entre le HDL et les lipoprotéines riches en triglycérides. 5 Un inhibiteur de la CETP, le torcetrapib, augmente les taux de HDL-C d’au moins 60%,9 mais entraîne un taux excessif de décès et d’événements cardio-vasculaires dans l’essai ILLUMINATE. 10 Bien que les raisons exactes de l'échec du torcetrapib demeurent incertaines, 11-16 l'étude des génotypes de la CETP peut aider à savoir si d'autres tentatives de prévention de la maladie coronarienne par inhibition de la CETP sont justifiées. Les études d'association génomique récentes montrent que les génotypes de la CETP sont associés aux taux de HDL-C plus fortement que tous les autres loci du génome. 17,18 En outre, alors qu'il peut être difficile de bien distinguer l'effet d'un agent pharmacologique sur la voie de la CETP de n'importe quel effet « hors cible » sur d'autres voies, des génotypes particuliers de la CETP devraient avoir des influences plus spécifiques, notamment sur la masse et l'activité de la CETP.
Une méta-analyse antérieure de 10 études 19 sur l'association entre la CETP et le risque coronaire s’était centrée sur 1 seul génotype de la CETP pour un total de 13 677 participants, dont 2857 cas coronaires. La ré-analyse actuelle des associations de 6 génotypes de la CETP avec les phénotypes de la CETP, les taux de lipides circulant, et avec le risque coronaire utilise les approches suivantes pour optimiser la puissance et pour réduire au minimum les biais : (1) nous rapportons des méta-analyses mises à jour sur la relation génotypes de la CETP, masse et activité de la CETP, HDL-C, cholestérol des lipoprotéines de basse densité (LDL-C), triglycérides, et apolipoprotéines AI et B (concernant des données sur 113 833 participants de 92 études) ; (2) nous rapportons une mise à jour des méta-analyses sur la relation entre génotypes de la CETP et critères coronaires (comportant des données sur 27 196 cas coronaires et 55 338 témoins de 46 études), incluant des données tabulaires cherchées auprès des investigateurs pour compléter et mettre à jour les données publiées; (3) nous avons contacté les principaux investigateurs d’études génétiques plus importantes d’association de variants autres que la CETP pour chercher les données non rapportées ; et (4) nous avons comparé l'association entre les augmentations génétiquement médiées des concentrations de HDL-C et le risque coronaire avec celles prévues en cas d’augmentation équivalente des concentrations de HDL-C dans les études prospectives disponibles.
METHODES
Sélection des études
Nous avons cherché les études publiées entre janvier 1970 et janvier 2008 sur les associations entre les génotypes de la CETP (numéro d'accession de GenBank NM_000078) et la masse et l'activité de la CETP, les concentrations de HDL-C, de LDL-C, des triglycérides, ou des apolipoprotéines AI et B, ou le risque d'infarctus du myocarde (défini par la surveillance multinationale de l'Organisation Mondiale de la Santé des tendances et des causes déterminantes dans les critères de maladie cardio-vasculaire [MONICA]) 20 ou les sténoses coronaires angiographiques (définies comme 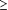 50% d'artères coronaires ou 50% d'artères coronaires ou 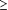 1 artère coronaire majeure). Pour les marqueurs des lipides, les données ont été employées seulement chez des personnes apparemment en bonne santé (personnes sans maladies coronariennes connues ou anomalies cliniques coronaires ou autres troubles des lipides) ayant des données au moins sur 1 génotype approprié. Les recherches électroniques, non limitées à l'anglais, ont été faites sur MEDLINE, EMBASE, BIOS, Science Citation Index, et la Chinese National Knowledge Infrastructure Database, et complétées en analysant les bibliographies des articles identifiés pour toutes les études appropriées et les articles synoptiques (y compris de méta-analyses), en faisant des recherches manuelles dans des journaux appropriés, et en correspondant avec les auteurs des études incluses. Les recherches sur ordinateur ont combiné la recherche de termes liés au génotype de la CETP (cholesteryl ester transfer protein, cholesterol ester transfer protein, CETP, gene, genes, loci, polymorphi*, allel*, phenotyp*, SNP*, RFLP*, chromosom*, variant, mutat*), phénotypes des lipides (HDL, LDL, triglycerides, apolipoproteins), et de la maladie coronarienne (myocardial infarction, atherosclerosis, coronary heart disease, coronary stenosis) sans restriction de langage (FIGURE 1). 1 artère coronaire majeure). Pour les marqueurs des lipides, les données ont été employées seulement chez des personnes apparemment en bonne santé (personnes sans maladies coronariennes connues ou anomalies cliniques coronaires ou autres troubles des lipides) ayant des données au moins sur 1 génotype approprié. Les recherches électroniques, non limitées à l'anglais, ont été faites sur MEDLINE, EMBASE, BIOS, Science Citation Index, et la Chinese National Knowledge Infrastructure Database, et complétées en analysant les bibliographies des articles identifiés pour toutes les études appropriées et les articles synoptiques (y compris de méta-analyses), en faisant des recherches manuelles dans des journaux appropriés, et en correspondant avec les auteurs des études incluses. Les recherches sur ordinateur ont combiné la recherche de termes liés au génotype de la CETP (cholesteryl ester transfer protein, cholesterol ester transfer protein, CETP, gene, genes, loci, polymorphi*, allel*, phenotyp*, SNP*, RFLP*, chromosom*, variant, mutat*), phénotypes des lipides (HDL, LDL, triglycerides, apolipoproteins), et de la maladie coronarienne (myocardial infarction, atherosclerosis, coronary heart disease, coronary stenosis) sans restriction de langage (FIGURE 1).
|
|

|
Figure 1. Distribution de l’étude
CETP correspond à Cholesteryl Ester Transfer Protein.
a Ces études tendant à être plus petites, elles ont comporté seulement un total d’environ 3% du nombre global de cas coronariens inclus dans cette revue, et seulement un total d’environ 8% du nombre global des participants inclus dans l'analyse des concentrations en lipides.
|
|
|
Recueil des données
Les informations ont été enregistrées pour TaqIB (rs708272), I405V (rs5882), –629C>A (rs1800775), D442G (rs2303790), –631C>A (rs1800776), et R451Q (rs1800777). Les données suivantes ont été extraites indépendamment par 3 investigateurs (S.E., D.S., et Z.Y.), employant un protocole précédemment décrit 21 : fréquences des génotypes suivant les critères catégoriels des maladie ; moyenne et écart-type des marqueurs des lipides suivant le génotype ; âge moyen des cas ; proportions d’hommes et de sous-groupes ethniques (définis par des personnes d'ascendance continentale européenne blanche, de l'Est Asiatique, ou autre [Asie du Sud, Moyen-Orient, Sud-Américain, et Nord-Africains]) ; le statut à jeun ou non et les méthodes de test. Les désaccords étaient résolus par discussion et par adjudication d'un quatrième auteur (A.T.). Nous avons employé les informations les plus à jour dans les cas de publications multiples. Nous avons complété les données publiées par une demande de données tabulaires (1) aux auteurs des études publiées, (2) aux investigateurs de 75 études non publiées mais potentiellement appropriées ayant inclus au moins 500 cas de maladie coronaire ou au moins 1000 participants en bonne santé énumérés dans les méta-analyses publiées 21-25 sur d’autres variants que ceux de la CETP, et (3) aux auteurs d’études d’association génomique larges ayant des critères appropriés.26,27
Analyse statistique
Les analyses ont été effectuées par l'utilisation de comparaisons dans l’étude pour limiter les biais possibles, prenant en compte les études qui avaient employé des méthodes admises et vérifiées de génotypage, d’analyse des lipides et de critères coronaires (tel que décrit ci-dessus). Les principales analyses étaient pré-spécifiées pour les modèles génétiques codominants. Les rapports récapitulatifs de cotes (OR) de maladie coronarienne et les taux moyens des marqueurs lipidiques (et les différences des taux moyens en comparaison des homozygotes communs) ont été calculés par l'utilisation d’un modèle des effets aléatoires qui incluait l'hétérogénéité inter-étude (avec des analyses de sensibilité utilisant des modèles à effets fixes). Pour permettre une comparaison de l'importance des associations avec plusieurs marqueurs lipidiques et phénotypes de la CETP, les associations ont été également présentées sous la forme de différences de pourcentage per-allèle (calculées en référence au taux moyen pondéré de chaque marqueur chez les homozygotes communs). Pour les études ayant comparé un seul groupe témoin à la fois avec l'infarctus du myocarde et les cas de sténose coronaire (sans chevauchement), nous avons évité un double comptage en analysant séparément l'infarctus du myocarde et les cas de sténose coronaire avant de les combiner dans un seul groupe maladie coronarienne. L'uniformité des résultats dans les études a été évaluée à l’aide d’un test statistique I2.28 L'hétérogénéité a été évaluée à l’aide d’un test statistique Q et en examinant les groupes pré-spécifiés des caractéristiques de l’étude. La preuve d’un biais de publication a été recherchée à l’aide d’un funnel plot, le test d'Egger, 29 et en comparant les résultats regroupés des études ayant au moins 500 cas coronaires (ou 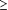 1000 participants en bonne santé pour les investigations gène-lipides) avec des résultats regroupés d’études plus petites. La preuve d’une déviation de l'équilibre Hardy-Weinberg a été évaluée par des tests du chi-2 utilisant les fréquences des génotypes chez les personnes en bonne santé, avec des analyses de sensibilité à des niveaux de signification de P<0.05 et de P<0.01. Les estimations du risque de maladie cardiaque des Prospective Studies Collaboration1 ont été utilisées pour calculer les OR attendus de maladie coronarienne correspondant aux augmentations per-allèle des taux du HDL-C observés avec les génotypes de la CETP au cours de cette revue, avec un test chi-2 pour l'uniformité des associations génotype- risque coronaire avec les associations risque coronaire-HDL-C. Toutes les analyses ont été faites en employant la version 10 de Stata (StataCorp LP, College Station, Texas). Tous les tests statistiques étaient bilatéraux à un niveau de signification de P<0.05, sauf indication contraire. 1000 participants en bonne santé pour les investigations gène-lipides) avec des résultats regroupés d’études plus petites. La preuve d’une déviation de l'équilibre Hardy-Weinberg a été évaluée par des tests du chi-2 utilisant les fréquences des génotypes chez les personnes en bonne santé, avec des analyses de sensibilité à des niveaux de signification de P<0.05 et de P<0.01. Les estimations du risque de maladie cardiaque des Prospective Studies Collaboration1 ont été utilisées pour calculer les OR attendus de maladie coronarienne correspondant aux augmentations per-allèle des taux du HDL-C observés avec les génotypes de la CETP au cours de cette revue, avec un test chi-2 pour l'uniformité des associations génotype- risque coronaire avec les associations risque coronaire-HDL-C. Toutes les analyses ont été faites en employant la version 10 de Stata (StataCorp LP, College Station, Texas). Tous les tests statistiques étaient bilatéraux à un niveau de signification de P<0.05, sauf indication contraire.
RESULTATS
Au total, 102 études appropriées incluant 147 599 individus ont été identifiées, dont 38 études avec des données tabulaires supplémentaires ou précédemment non publiées (Figure 1, et eTable 1, et eTable 2, disponibles sur www.jama.com).17,30-165 Quarante-cinq études provenaient d’Europe, 15 d’Amérique du Nord, 29 d’Extrême-Orient (principalement Chine et Japon), 11 d'autres régions, et 2 étaient multinationales. Vingt et une études étaient prospectives et 81 étaient transversales ou cas-témoins. La fréquence des allèles mineurs chez les individus blancs en bonne santé était de 0.42 pour TaqIB (rs708272), 0.35 pour I405V (rs5882), 0.48 pour –629C>A (rs1800775), moins de 0.01 pour D442G (rs2303790), 0.08 pour –631C>A (rs1800776), et 0.04 pour R451Q (rs1800777) (TABLEAU 1). Les études précédentes ont indiqué un linkage presque complet de TaqIB avec –629C>A, mais les autres génotypes de la CETP pris en compte dans cette revue semblent être seulement faiblement corrélés.166
|
|

|
Tableau 1. Description des génotypes de la CETP inclus dans la revue
|
|
|
Phénotypes de la CETP et taux de lipides
Quatre-vingt-douze études sur les génotypes de la CETP ont été identifiées portant sur 113 833 personnes ayant rapporté la masse de la CETP, l'activité de la CETP, les taux de lipides circulant, ou les trois. 17,30-148 incluant 36 études fournissant des données supplémentaires ou précédemment non publiées sur 49 502 individus (TABLEAU 2). Parmi les 91 études ayant évalué les associations avec le HDL-C, 7 ont utilisé des méthodes de vérification homogènes pour mesurer les taux de HDL-C, 49 des méthodes non homogènes, et 35 n'ont pas rapporté la méthode de vérification utilisée. Globalement pour chaque allèle A transmis, les porteurs du variant TaqIB avaient une masse moyenne inférieure de la CETP (–9.7% ; intervalle de confiance à 95% [IC], –11.7% à –7.8%), une activité moyenne plus basse de la CETP (–8.6% ; IC 95%, –13.0% à –4.1%), des taux moyens plus élevés de HDL-C (4.5% ; IC 95%, 3.8%-5.2%), des taux moyens plus élevés d'apolipoprotéine AI (2.4% ; IC 95%, 1.6%-3.2%), des taux moyens plus faibles de LDL-C (–0.9% ; IC 95%, –1.6% à –0.3%), des taux moyens plus faibles d'apolipoprotéine B (–0.5% ; IC 95%, –1.1% à 0.1%), et des taux moyens plus bas de triglycérides (–2.0% ; IC 95%, –3.2% à –0.7%) que les homozygotes communs (Figure 2). Globalement, par allèle G transmis, les porteurs du variante I405V avaient une masse moyenne inférieure de la CETP (–5.7% ; IC 95%, –7.5% à –4.0%), une activité moyenne plus basse de CETP (–8.2% ; IC 95%, –17.8% à 1.3%), des taux moyens plus élevés de HDL-C (2.5% ; IC 95%, 1.8%-3.2%), des taux moyens plus élevés d'apolipoprotéine AI (1.6% ; IC 95%, 1.2%-2.0%), et des taux moyens plus bas de triglycérides (–2.1% ; IC 95%, –3.0% à –1.1%) que les homozygotes communs, sans différence perceptible du LDL-C ou des taux d'apolipoprotéine B. Globalement, par allèle A transmis, les porteurs du variant –629C>A avaient une masse moyenne inférieure de CETP (–9.4% ; IC 95%, –13.8% à –5.0%), une activité moyenne plus basse de CETP (–5.9% ; IC 95%, –11.0% à –0.8%), des taux moyens plus élevés de HDL-C (4.9% ; IC 95%, 4.3%-5.4%), des taux moyens plus élevés d'apolipoprotéine AI (1.8% ; IC 95%, 0.7% - 2.8%), et des taux moyens plus bas de triglycérides (–2.1% ; IC 95%, –3.4% à –0.9%) que les homozygotes communs mais sans différence perceptible du LDL-C ou des taux d'apolipoprotéine B. Les données étaient insuffisantes pour faire des estimations informatives par allèle pour D442G, –631C>A, et R451Q (Tableau 2) ; cependant, dans les modèles dominants, elles étaient associées à des différences moyennes du HDL-C respectivement de 13.4% (IC 95%, 9.4% - 17.4%), –0.7% (IC 95%, –2.4% à 1.0%), et –8.8% (IC 95%, –9.7% à –7.9%) comparées à celles des homozygotes communs.
|
|

|
Tableau 2. Résumé des données disponibles sur les génotypes de la CETP, les phénotypes de la CETP, les taux de lipides, et les critères coronariens a
|
|
|
|
|

|
Figure 2. Associations of CETP Genotypes With CETP Phenotypes and Lipid Levels
CETP correspond à Cholesteryl Ester Transfer Protein; IC, intervalle de confiance ; HDL-Ccholestérol des lipoprotéines de haute densité ; LDL-C, cholestérol des lipoprotéines de basse densité. Pour convertir les apolipoprotéines AI et B en mg/dl, diviser par 0.01 ; pour convertir le HDL-C et le LDL-C en mg/dl, diviser par 0.0259 ; et pour convertir les triglycécides en mg/dl, diviser par 0.0113. Évaluation d'hétérogénéité : I2 (IC à 95%) pour la masse de la CETP, l'activité de la CETP, le HDL-C, l'apolipoprotéine AI, le LDL-C, l'apolipoprotéine B, et les triglycérides, était respectivement de 66% (39%-81%), 71% (44%-86%), 75% (69%-80%), 66% (46%-78%), 51% (32%-65%), 14% (0%-51%), et 49% (30%-62%) pour TaqIB ; 0% (0%-71%), NA*, 56% (33%-71%), 0% (0%-68%), 24% (0%-58%), 16% (0%-60%), et 0% (0%-49%) pour I405V ; et 71% (17%-90%), NA*, 37% (0%-61%), 36% (0%-78%), 29% (0%-63%), 0% (0%-90%), et 0% (0%-57%) pour –629C>A. NA* indique que les statistiques I2 n'ont pas été calculées quand il y avait seulement 2 études.
a Les évaluations regroupées ont été calculées par des modèles d’effets aléatoires. Les évaluations calculées par des modèles à effet fixe sont montrées dans le eTableau 3, disponible sur www.jama.com.
b Différences moyennes normalisées.
c Calculé concernant le taux moyen pondéré de chaque marqueur chez les homozygotes communs.
|
|
|
Il y avait des preuves d'hétérogénéité dans les associations avec le HDL-C dans les études (TaqIB : I2 = 75% ; IC 95%, 69%-80% ; I405V : I2 = 56% ; IC 95%, 33%-71% ; –629C>A : I2 = 37% ; IC 95%, 0%-61%). Cette hétérogénéité était en partie expliquée par les caractéristiques de niveau des études enregistrées, y compris la population source (TaqIB et –629C>A), appartenance ethnique (TaqIB et –629C>A), la taille de l’étude (I405V), et données obtenues en correspondant avec des investigateurs ou extraites directement à partir des articles publiés (TaqIB) (Figure 3). Une déviation significative de l'équilibre Hardy-Weinberg (P<0.05) a été détectée dans 10 études pour TaqIB (5 études y compris à P<0.01), 2 études pour I405V (les deux études à P<0.01), et 4 études pour –629C>A (dont 2 études à P<0.01), mais leur exclusion n'a pas matériellement changé les résultats. Les analyses par taille d’étude (Figure 3) et les autres tests standards (eFigure disponible sur www.jama.com) n'ont pas montré de preuve irréfutable d’un biais de publication.
|
|

|
Figure 3. Différences moyennes des taux du HDL-C associées aux génotypes de la CETP, groupés selon les caractéristiques enregistrées de l’étude
CETP correspond à Cholesteryl Ester Transfer Protein; IC intervalle de confiance; HDL-C, cholestérol des lipoprotéines de haute densité. Pour convertir le HDL-C en mg/dl, diviser par 0.0259. Les tailles des marqueurs de données sont proportionnelles à l'inverse de la variance de la différence moyenne pondérée. Pour le sexe et l'appartenance ethnique, les études ont pu avoir des données de plus d’une catégorie. Des évaluations globales ont été calculées en employant des modèles d’effets aléatoires (les évaluations à effet fixe sont fournies dans le eTableau 3, disponible sur www.jama.com). Plusieurs caractéristiques enregistrées ont expliqué une partie de l'hétérogénéité observée, y compris l'appartenance ethnique (P=0.008), la source de population (P=0.04), et la source des données (P<0.001) pour TaqIB ; la taille de l’étude (P=0.02) pour I405V ; et l’appartenance ethnique (P<0.001) et la source de la population (P=0.007) pour –629C>A.
|
|
|
Résultats coronaires
Quarante-six études fournissaient des données sur 27 196 cas coronaires et 55 338 témoins,30-35, 39, 42-45, 51-53, 59, 61, 63-68, 71, 78-81, 85-87, 90, 94, 98-100, 103, 105, 107- 111, 113-115, 117-123, 133, 135, 138-140, 142, 143, 145-148, 150-165 dont 21 études ayant fourni des données supplémentaires ou précédemment non publiées sur 17 187 cas et 38 619 témoins. Trente-trois études ont recruté les témoins dans la population générale, 11 études ont recrutées leurs témoins dans des hôpitaux ou dans des essais cliniques, 2 études ayant employé les deux sources. Les OR combinés de maladie coronarienne était de 0.95 (IC 95%, 0.92-0.99) par allèle A du variant TaqIB. Il y avait une faible hétérogénéité potentielle dans les 38 études disponibles (I2=18% ; IC 95%, 0%-45% ; P=0.17), avec des résultats légèrement plus faibles dans les études plus importantes et dans les études ayant fourni des données par correspondance (P=0.01 et P=0.003, respectivement, pour l’hétérogénéité) (Figure 4). L’OR combiné de maladie coronarienne était de 0.94 (IC 95%, 0.89-1.00) par allèle G du variant I405V. Il y avait une faible hétérogénéité potentielle dans les 18 études disponibles (I2=39% ; IC 95%, 0%-66% ; P=0.04), expliquée pour la plupart par des différences dans le choix des groupes témoins (P<0.001). L’OR combiné de maladie coronarienne était de 0.95 (IC 95%, 0.91-1.00) par allèle A du variant –629C>A. Il y avait une hétérogénéité modérée potentielle dans les 17 études disponibles (I2=32% ; IC 95%, 0%-62% ; P=0.10), dont peu était expliqué par les caractéristiques des études enregistrées.
|
|

|
Figure 4. Génotypes de la CETP et risque coronaire, groupés selon les caractéristiques enregistrées de l’étude
CETP correspond à Cholesteryl Ester Transfer Protein; IC, intervalle de confiance. Les tailles des marqueurs des données sont proportionnelles à l'inverse de la variance du log e du rapport de cotes. Pour l'appartenance ethnique, la source des témoins, et les critères évalués, les études ont pu avoir des données de plus d’une catégorie. Pour l'appartenance ethnique, les résultats ne sont pas présentés pour 4 études de TaqIB et 2 études d'I405V et de –629C>A principalement basées chez des non blancs, des Asiatiques en dehors de l’Extrême Orient. Pour les résultats évalués dans TaqIB, les résultats ne sont pas présentés pour une étude qui n'a pas fourni les fréquences de génotype séparément pour les cas d'infarctus du myocarde et de sténose coronaire. Évaluation d'hétérogénéité : TaqIB (I2=18% ; IC 95%, 0%-45%), I405V (I2=39% ; IC 95%, 0%-66%), ou –629C>A (I2=32% ; IC 95%, 0%-62%). L'hétérogénéité observée pourrait être partiellement expliquée par la taille d'étude (P=0.01) et la source des (P=0.003) pour TaqIB et par la source des témoins (P<0.001) pour I405V (autres comparaisons P<0.05 pour chacun). Les évaluations globales ont été calculées en employant des modèles à effets aléatoires; ceux calculés en utilisant des modèles à effet fixe étaient de 0.96 (IC 95%95%, 0.93-0.99) pour TaqIB, 0.95 (IC 95%, 0.92 - 0.99) pour I405V, et 0.95 (IC 95%, 0.91-0.99) pour –629C>A.
|
|
|
Une déviation significative de l'équilibre Hardy-Weinberg (P<0.05) a été détectée chez les témoins de 6 études pour TaqIB (dont 2 études à P<0.01), d’une étude pour I405V (P<0.01), et de 3 études pour –629C>A (dont 1 étude à P<0.01). L'exclusion de ces dernières études n'a pas matériellement changé les résultats. Ceci a été le cas pour les études des phénotypes de la CETP et des taux de lipides, il n'y a pas eu de preuve de biais de publication dans les tests standards (eFigure), à l'exception possible de TaqIB (Figure 4). Les données étaient insuffisantes pour fournir des informations du risque pour les 3 génotypes rares de la CETP. La Figure 5 montre les estimations des associations entre le HDL-C et le risque coronaire dérivés de Prospective Studies Collaboration, 1 avec une comparaison des OR pour les augmentations de HDL-C par allèle du risque de maladie coronarienne dans les études génétiques comparées aux OR dans les études prospectives sur le HDL-C (pour TaqIB, 0.95 ; IC 95%, 0.92-0.99 contre 0.92 ; IC 95%, 0.91-0.93 ; P pour l’hétérogénéité=0.11 ; pour I405V, 0.94 ; IC 95%, 0.89-1.00 contre 0.95 ; IC 95%, 0.94-0.97 ; P=0.72 ; et pour –629C>A, 0.95 ; IC 95%, 0.91-1.00 versus 0.92 ; IC 95%, 0.91-0.93 ; P=0.19).
|
|

|
Figure 5. Les rapports observés de cotes par allèle pour la maladie coronarienne avec les variants de la CETP versus rapports de cotes sont dérivés des études prospectives disponibles sur les taux de HDL-C
CETP correspond à Cholesteryl Ester Transfer Protein ; IC, intervalle de confiance; HDL-C, cholestérol des lipoprotéines de haute densité. Les tailles des marqueurs de données sont proportionnelles à l'inverse de la variance de l'évaluation du log e de l’estimation du risque. Test du chi-2 pour la différence : P=0.11 pour TaqIB, P=0.72 pour I405V, et P=0.19 pour –629C>A.
a rapport de cotes par allèle de maladie coronarienne suivant les indications de la Figure 4.
b rapports de risque de maladie coronarienne calculée en employant les évaluations de risque de 153 798 participants de 61 études 1 pour une augmentation des taux habituels de HDL-C égaux à ceux observés par allèle pour TaqIB, I405V, et –629C>A (Figure 2).
|
|
|
COMMENTAIRE
L'étude des génotypes de la CETP qui modifient la masse et l'activité de celle-ci peut aider à clarifier l’importance de la voie de la CETP dans le risque métabolique lipidique et de maladie coronarienne. Cependant, certains génotypes particuliers de la CETP étant faiblement associés aux phénotypes de la CETP et aux taux des lipides, des études génétiques fiables peuvent exiger des milliers de participants, dont un grand nombre de patients présentant une maladie coronarienne. En l'absence d’études portant sur un grand nombre d’individus, nous avons conduit une méta-analyse mise à jour sur un total de plus de 147 000 personnes (dont plus de 27 000 cas coronariens). Nous avons démontré que des génotypes communs de la CETP diminuent la masse et l'activité de celle-ci d’environ 5% à 10%, augmentent le HDL-C et l'apolipoprotéine AI d’environ 3% à 5% (ce qui est comparable aux différences observées du HDL-C entre fumeurs et non-fumeurs), et diminuent les triglycérides d’environ 2%. Les associations avec le LDL-C et l’apolipoprotéine B étaient encore plus faibles ou négligeables. Nous avons prouvé que les mêmes génotypes de la CETP qui sont faiblement associés à des taux élevés de HDL-C sont associés faiblement et inversement au risque coronaire. L'importance de la réduction du risque par allèle était compatible avec celle attendue sur la base des associations observées entre les taux de HDL-C et le risque coronaire dans les études prospectives (Figure 5).1 Comme nous le discutons ci-dessous cependant, la quantité et la qualité des données génétiques disponibles exigent un examen soigneux.
Comparée à une précédente méta-analyse19 qui ne s’était concentrée que sur les associations du variant TaqIB avec les taux de HDL-C et le risque de maladie coronarienne, notre revue prend en compte 6 génotypes de la CETP, évalue les associations avec plusieurs différents marqueurs lipidiques, et concerne 10 fois plus de données. Ces données fournissent une plus grande puissance que précédemment disponible et doivent réduire la possibilité de biais de publication. 21 Cependant, bien que nous n'ayons pas détecté de preuve irréfutable de biais de publication, il est difficile de ne pas tenir compte d’un biais, en particulier en raison des faibles associations observées et la fiabilité de ces associations résulte du regroupement des études plus larges et plus petites, et de la faible sensibilité globale des tests statistiques pour les biais de publication. (Dans le meilleur des cas, en disposant de nombres plus importants, les analyses regroupées ne concerneraient les données que des larges études, pouvant être moins exposées à des biais de publication.) Comme nous n'avons pas eu accès aux données individuelles, nous n’avons pas pu contrôler sur la stratification des populations, 167 conduire des analyses mendéliennes de randomisation, 168,169 ajuster sur les variables des voies intermédiaires possibles, explorer l'hétérogénéité selon les caractéristiques des niveaux individuels, ou réaliser des analyses d’haplotype. Ces considérations accentuent le besoin d’études larges sur les génotypes de la CETP avec une évaluation concomitante des phénotypes de la CETP et des taux des lipides, impliquant peut-être plus de 10 000 cas coronariens et un nombre semblable de témoins. En outre, les études à grande échelle avec des données sur le style de vie sont nécessaires pour explorer les effets communs potentiels des génotypes de la CETP avec les causes déterminantes environnementales des taux de HDL-C (par exemple, exercice et alcool) sur le risque de maladie coronarienne. Même des études plus importantes seront nécessaires pour évaluer de façon fiable les génotypes moins communs de la CETP.170
Contrairement aux preuves disponibles des épreuves randomisées à court terme sur l’inhibition de la CETP,10,14 nos analyses suggèrent que les individus ayant des taux accrus de HDL-C (vraisemblablement permanents) en raison de diminutions génétiques de la CETP peuvent avoir un risque coronaire légèrement réduit. Cette anomalie apparente peut être due aux effets « hors cible » potentiellement spécifiques au torcetrapib (notamment l’interférence avec le système rénine angiotensine et l’élévation de la pression artérielle). 10 D’un autre côté, il a été suggéré que les particules de HDL produites dans les conditions d'inhibition de la CETP pourraient être dysfonctionnelles, n'importe quelle augmentation apparente des taux de HDL-C étant compensée par la clairance compensatrice du HDL-C par la voie hépatique directe 11 et la réduction du déplacement médiée par l’apolipoprotéine A-1 du cholestérol libre intracellulaire des macrophages. 11,12 D’autres études sur d’autres inhibiteurs de la CETP 171 et des investigations sur les génotypes de la CETP en relation avec la pression artérielle et à d'autres traits peuvent aider à expliquer ces questions mécanistiques.
CONCLUSION
En résumé, 3 génotypes de la CETP sont associés à une inhibition modérée de l'activité de la CETP, à des taux légèrement plus élevés du HDL-C, et à une faible inversion du risque coronaire. Cette étude illustre le besoin d’études plus importantes pour démontrer l’impact modeste que les variants génétiques simples ont sur des résultats complexes tels que la maladie coronarienne. D'autres études sont justifiées pour déterminer la valeur de l'inhibition de la CETP dans la prévention de la maladie coronarienne.
Informations sur les auteurs
Correspondance: John Danesh, DPhil, FRCP, Department of Public Health and Primary Care, University of Cambridge, Strangeways Research Laboratory, Worts Causeway, Cambridge, England CB1 8RN (john.danesh{at}phpc.cam.ac.uk).
Contributions des auteurs : Le Dr. Danesh et M. Thompson ont eu un accès complet à toutes les données de l'étude et acceptent la responsabilité de l'intégrité des données et de l'exactitude de l'analyse des données.
Conception et schéma de l’étude: Thompson, Di Angelantonio, Sarwar, Dullaart, Ye, Danesh.
Recueil des données: Thompson, Di Angelantonio, Sarwar, Erqou, Saleheen, Dullaart, Ye, Danesh.
Analyse et interprétation des données: Thompson, Di Angelantonio, Sarwar, Erqou, Keavney, Ye, Danesh.
Rédaction du manuscrit: Thompson, Di Angelantonio, Sarwar, Danesh.
Revue critique du manuscrit: Thompson, Di Angelantonio, Sarwar, Erqou, Saleheen, Dullaart, Keavney, Ye, Danesh.
Analyse statistique: Thompson, Di Angelantonio, Sarwar, Erqou.
Obtention du financement: Danesh.
Aide administrative, technique, ou matérielle: Thompson, Di Angelantonio, Erqou, Saleheen, Dullaart, Ye, Danesh.
Supervision de l’étude: Danesh. Drs Di Angelantonio et Erqou, et Messrs Thompson et Sarwar ont contribute à parts égales à cet article et sont considérés comme premiers co-auteurs.
Liens financiers: Aucun déclaré.
Financement/Soutien: Ce travail a bénéficié du soutien sous la forme d’une bourse du programme de la British Heart Foundation. Le Dr. Danesh a bénéficié du soutien sous la forme d’un prix Raymond and Beverly Sackler Award in Medical Sciences. Le Dr. Di Angelantonio et M. Thompson ont bénéficié du soutien ainsi que M. Sarwar d’une bourse de doctorat UK Medical Research Council PhD studentship, le Dr. Erqou d’une bourse Gates Cambridge Scholarship et the UK Overseas Research Trust. Le Dr. Saleheen a bénéficié du soutien de la Yousef Jameel Foundation. Certains côtés de ce travail ont bénéficié du soutien sous la forme d’une bourse éducationnelle de GlaxoSmithKline.
Rôle des sponsors: Les commanditaires n'ont joué aucun rôle dans la conception et la conduite de l'étude, dans le recueil, la gestion, l'analyse, et l'interprétation des données, ou dans la préparation, la revue, ou l'approbation du manuscrit.
Autres informations: La eFigure et les eTableaux 1-3 sont disponibbles à http://www.jama.com.
Autres contributions: Rory Collins, FMedSci, FRCP (University of Oxford, Oxford, England), ont apporté des commentaires précieux. Angela Harper (University of Cambridge, Cambridge, England) a fourni un soutien administratif. Les investigateurs suivants ont fourni des informations utiles sur leurs études: Birgit Agerholm-Larsen, MSc, PhD, Herlev University Hospital, Herlev, Denmark; Nassr Al-Daghri, MD, Birmingham Heartlands Hospital, Birmingham, England; Rolf V. Andersen, MSc, PhD, Department of Clinical Biochemistry, Herlev University Hospital, Herlev, Denmark; Yasumichi Arai, PhD, Keio University School of Medicine, Tokyo, Japan; Gil Atzmon, PhD, Albert Einstein College of Medicine, New York, New York; Nir Barzilai, MD, Albert Einstein College of Medicine, New York, New York; Anja Bauerfeind, PhD, Humboldt University of Berlin, Berlin, Germany; Susanna E. Borggreve, MD, University Medical Center Groningen, Groningen, the Netherlands;Michiel L. Bots, MD, PhD, Julius Centre for Health Sciences and Primary Care, University Medical Centre Utrecht, Utrecht, the Netherlands; Hannia Campos, PhD, Harvard School of Public Health, Boston, Massachusetts; Peter Clifton, MD, PhD, CSIRO Human Nutrition, Adelaide, Australia; Eliana C. de Faria, MD, MS, PhD, State University of Campinas, Sao Paulo, Brazil; Robin P. F. Dullaart (au nom du PREVEND Study Group), MD, PhD, University Medical Center Groningen, Groningen, the Netherlands; Moses Elisaf, MD, University of Ioannina, Ioannina, Greece; Jeanette Erdmann, PhD, Medizinische Klinik II, Lübeck University, Lübeck, Germany; Dilys Freeman, PhD, University of Glasgow, Glasgow, Scotland; Domenico Girelli, MD, PhD, University of Verona, Verona, Italy; Akitomo Goto, MD, Nagoya City University, Nagoya, Japan; John Griffin, PhD, The Scripps Research Institute, La Jolla, California; Wendy Hall, PhD, King’s College, London, England; Mohamed Hammami, MD, Monastir University, Monastir, Tunisia; A. Geert Heidema, MSc, University of Maastricht, Maastricht, the Netherlands; BenjaminD. Horne (on behalf of the Intermountain Heart Collaborative Study Group), PhD, MPH, Intermountain Medical Center, Murray, Utah; Akihiro Inazu, MD, PhD, Kanazawa University, Kanazawa, Japan; Aaron Isaacs, DSc, Genetic Epidemiology Unit, Erasmus MC University Medical Center, Rotterdam, the Netherlands; Turgay Isbir, MD, University of Istanbul, Istanbul, Turkey; Yangsoo Jang, MD, PhD, Division of Cardiology, Cardiovascular GenomeCenter, Yonsei Medical Institute, Yonsei University, Seoul, South Korea; Majken K. Jensen, MSc, Department of Nutrition, Harvard School of Public Health, Boston, Massachusetts; Bernard Keavney, MD, FRCP (on behalf of the ISIS Collaborative Group), Newcastle University, Newcastle, England; Kathy Klos, PhD, University of Texas–Houston Health Science Center, Houston, Texas; Jong Ho Lee, PhD, Yonsei University Research Institute of Science for Aging, Yonsei University, Seoul, South Korea; Robert W.Mahley,MD,PhD,University of California, San Francisco; Massimo Mangino, PhD, University of Leicester, Leicester, England; Nicola Martinelli, MD, University of Verona, Verona, Italy; Pamela McCaskie, BSc(Hons), University of Western Australia, Perth, Australia; Manjari Mukherjee, MD, India N.H. Hospital, Bangalore, India; Børge G. Nordestgaard, MD, DMSc, Herlev University Hospital, Herlev, Denmark; Kenji Okumura,MD,Nagoya University School of Medicine, Nagoya, Japan; Oliviero Olivieri, MD, University of Verona, Verona, Italy; Natalie Pecheniuk, PhD, The Scripps Research Institute, La Jolla, California; Jogchum Plat, PhD, Maastricht University, Maastricht, the Netherlands; QinQin, MD, Tianjin Chest Hospital, Tianjin, China; Eric B. Rimm, ScD, Department of Epidemiology, Harvard School of Public Health, Boston, Massachusetts; Nilesh Samani, MD, FRCP, University of Leicester, Leicester, England; José V. Sorli, D,University of Valencia, Valencia, Spain; John F. Thompson, PhD, Pfizer Global Research and Development, Groton, Connecticut; Martin Tobin, PhD, University of Leicester, Leicester, England; Anne Tybjærg- Hansen, MD, DMSc, Copenhagen University Hospital, Copenhagen, Denmark; Yvonne van der Schouw, PhD, Julius Centre forHealth Sciences and Primary Care, University Medical Centre Utrecht, Utrecht, the Netherlands; Cornelia M. van Duijn, PhD, Genetic Epidemiology Unit, Erasmus MC University Medical Center, Rotterdam, the Netherlands; Mitsuhiro Yokota, MD, PhD, FACC, Aichi-Gakuin University, Nagoya, Japan; Shinji Yokoyama, MD, PhD, FRCPC, Nagoya City University, Nagoya, Japan; Mohammad Hadi Zafarmand, MD, Julius Centre for Health Sciences and Primary Care, University Medical Centre Utrecht, Utrecht, the Netherlands; Guo-Bing Zhang, MD, Shanghai First People’s Hospital, Shanghai, China. Aucune de ces personnes n’a reçu de compensation financière.
Affiliations des auteurs : Department of Public Health and Primary Care, University of Cambridge, Cambridge, England (Drs Di Angelantonio, Erqou, Saleheen, Ye, Danesh, et Messrs Thompson et Sarwar); Department of Endocrinology, University Medical Center Groningen, University of Groningen, Groningen, Pays Bas (Dr Dullaart); Institute of Human Genetics, Newcastle University, Newcastle, England (Dr Keavney).
Pour le commentaire éditorial voir p 2795.
FMC disponible en ligne à www.jamaarchivescme.com et questions p 2804.
BIBLIOGRAPHIE
 |  |
1. Prospective Studies Collaboration.; Lewington S, Whitlock G, Clarke R, et al. Blood cholesterol and vascular mortality by age, sex, and blood pressure: a metaanalysis of individual data from 61 prospective studies with 55,000 vascular deaths. Lancet. 2007;370(9602): 1829-1839.
PUBMED
2. Packard C.. A triumvirate of targets in the prevention and treatment paradigm for cardiovascular disease. Atheroscler Suppl. 2006;7(1):21-29.
PUBMED
3. Singh IM, Shishehbor MH, Ansell BJ. High-density lipoprotein as a therapeutic target: a systematic review. JAMA. 2007;298(7):786-798.
FREE FULL TEXT
4. Nicholls SJ, Tuzcu EM, Sipahi I, et al. Statins, highdensity lipoprotein cholesterol, and regression of coronary atherosclerosis. JAMA. 2007;297(5):499-508.
FREE FULL TEXT
5. Hamilton JA, Deckelbaum RJ. Crystal structure of CETP: new hopes for raising HDL to decrease risk of cardiovascular disease? Nat Struct Mol Biol. 2007;14 (2):95-97.
PUBMED
6. Packard C, Nunn A, Hobbs R. High-density lipoprotein: guardian of the vascular system? Int J Clin Pract. 2002;56(10):761-771.
PUBMED
7. Inazu A, Brown ML, Hesler CB, et al. Increased highdensity lipoprotein levels caused by a common cholesteryl-ester transfer protein gene mutation. N Engl J Med. 1990;323(18):1234-1238.
PUBMED
8. Okamoto H, Yonemori F, Wakitani K, et al. A cholesteryl ester transfer protein inhibitor attenuates atherosclerosis in rabbits. Nature. 2000;406(6792): 203-207.
PUBMED
9. Clark RW, Sutfin TA, Ruggeri RB, et al. Raising highdensity lipoprotein in humans through inhibition of cholesteryl ester transfer protein: an initial multidose study of torcetrapib. Arterioscler Thromb Vasc Biol. 2004; 24(3):490-497.
FREE FULL TEXT
10. Barter PJ, Caulfield M, Eriksson M, et al.; ILLUMINATE Investigators. Effects of torcetrapib in patients at high risk for coronary events. N Engl J Med. 2007;357 (21):2109-2122.
PUBMED
11. Tall AR. CETP inhibitors to increase HDL cholesterol levels. N Engl J Med. 2007;356(13):1364-1366.
PUBMED
12. Tall AR, Yvan-Charvet L, Wang N. The failure of torcetrapib: was it the molecule or the mechanism? Arterioscler Thromb Vasc Biol. 2007;27(2):257-260.
FREE FULL TEXT
13. Dullaart RP, Dallinga-Thie GM, Wolffenbuttel BH, van Tol A. CETP inhibition in cardiovascular risk management: a critical appraisal. Eur J Clin Invest. 2007; 37(2):90-98.
PUBMED
14. Bots ML, Visseren FL, Evans GW, et al. Torcetrapib and carotid intima-media thickness in mixed dyslipidaemia (RADIANCE 2 study): a randomised, doubleblind trial. Lancet. 2007;370(9582):153-160.
PUBMED
15. Mazzone T.. HDL cholesterol and atherosclerosis. Lancet. 2007;370(9582):107-108.
PUBMED
16. Rader DJ. Illuminating HDL: is it still a viable therapeutic target? N Engl J Med. 2007;357(21):2180- 2183.
PUBMED
17. Kathiresan S, Melander O, Guiducci C, et al. Six new loci associated with blood low-density lipoprotein cholesterol, high-density lipoprotein cholesterol or triglycerides in humans. Nat Genet. 2008;40(2):189-197.
PUBMED
18. Willer CJ, Sanna S, Jackson AU, et al. Newly identified loci that influence lipid concentrations and risk of coronary artery disease. Nat Genet. 2008;40(2): 161-169.
PUBMED
19. Boekholdt SM, Sacks FM, Jukema JW, et al. Cholesteryl ester transfer protein TaqIB variant, highdensity lipoprotein cholesterol levels, cardiovascular risk, and efficacy of pravastatin treatment: individual patient meta-analysis of 13,677 subjects. Circulation. 2005; 111(3):278-287.
FREE FULL TEXT
20. Tunstall-Pedoe H, Kuulasmaa K, Amouyel P, et al. Myocardial infarction and coronary deaths in the World Health Organization MONICA Project: registration procedures, event rates, and case-fatality rates in 38 populations from 21 countries in four continents. Circulation. 1994;90(1):583-612.
FREE FULL TEXT
21. Bennet AM, Di Angelantonio E, Ye Z, et al. Association of apolipoprotein E genotypes with lipid levels and coronary risk. JAMA. 2007;298(11):1300-1311.
FREE FULL TEXT
22. Casas JP, Cavalleri GL, Bautista LE, et al. Endothelial nitric oxide synthase gene polymorphisms and cardiovascular disease: a HuGE review. Am J Epidemiol. 2006;164(10):921-935.
FREE FULL TEXT
23. Lewis SJ, Ebrahim S, Davey Smith G. Meta- analysis of MTHFR 677C-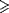 T polymorphism and coronary heart disease: does totality of evidence support causal role for homocysteine and preventive potential of folate? BMJ. 2005;331(7524):1053.
FREE FULL TEXT
24. Wheeler JG, Keavney BD, Watkins H, Collins R, Danesh J. Four paraoxonase gene polymorphisms in 11212 cases of coronary heart disease and 12786 controls: meta-analysis of 43 studies. Lancet. 2004; 363(9410):689-695.
PUBMED
25. Ye Z, Liu EH, Higgins JP, et al. Seven haemostatic gene polymorphisms in coronary disease: metaanalysis of 66,155 cases and 91,307 controls. Lancet. 2006;367(9511):651-658.
PUBMED
26. Samani NJ, Erdmann J, Hall AS, et al. Genomewide association analysis of coronary artery disease. N Engl J Med. 2007;357(5):443-453.
PUBMED
27. Wellcome Trust Case Control Consortium. Genomewide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 2007; 447(7145):661-678.
PUBMED
28. Higgins JP, Thompson SG, Deeks JJ, Altman DG. Measuring inconsistency in meta-analyses. BMJ. 2003; 327(7414):557-560.
FREE FULL TEXT
29. Egger M, Davey Smith G, Schneider M, Minder C. Bias in meta-analysis detected by a simple, graphical test. BMJ. 1997;315(7109):629-634.
FREE FULL TEXT
30. Thompson JF, Wood LS, Pickering EH, Dechairo B, Hyde CL. High-density genotyping and functional SNP localization in the CETP gene. J Lipid Res. 2007; 48(2):434-443.
FREE FULL TEXT
31. Thompson JF, Durham LK, Lira ME, Shear C, Milos PM. CETP polymorphisms associated with HDL cholesterol may differ from those associated with cardiovascular disease. Atherosclerosis. 2005;181(1): 45-53.
PUBMED
32. Lloyd DB, Lira ME, Wood LS, et al. Cholesteryl ester transfer protein variants have differential stability but uniform inhibition by torcetrapib. J Biol Chem. 2005; 280(15):14918-14922.
FREE FULL TEXT
33. Hinds DA, Seymour AB, Durham LK, et al. Application of pooled genotyping to scan candidate regions for association with HDL cholesterol levels. Hum Genomics. 2004;1(6):421-434.
PUBMED
34. Lira ME, Lloyd DB, Hallowell S, Milos PM, Thompson JF. Highly polymorphic repeat region in the CETP promoter induces unusual DNA structure. Biochim Biophys Acta. 2004;1684(1-3):38-45.
PUBMED
35. Thompson JF, Lloyd DB, Lira ME, Milos PM. Cholesteryl ester transfer protein promoter singlenucleotide polymorphisms in Sp1-binding sites affect transcription and are associatedwith high-density lipoprotein cholesterol. Clin Genet. 2004;66(3):223-228.
PUBMED
36. Akita H, Chiba H, Tsuchihashi K, et al. Cholesteryl ester transfer protein gene: two commonmutations and their effect on plasma high-density lipoprotein cholesterol content. J Clin Endocrinol Metab. 1994;79 (6):1615-1618.
ABSTRACT
37. Al-Daghri NM, Al-Attas O, Patel A, et al. Association between the cholesteryl ester transfer protein TaqIdetectable B polymorphism and low high-density lipoprotein cholesterol concentration in Saudis. Clin Sci (Lond). 2003;105(4):467-472.
PUBMED
38. Arai Y, Hirose N, Yamamura K, et al. Deficiency of choresteryl ester transfer protein and gene polymorphisms of lipoprotein lipase and hepatic lipase are not associated with longevity. J Mol Med. 2003;81(2): 102-109.
PUBMED
39. Arca M, Montali A, Ombres D, et al. Lack of association of the common TaqIB polymorphism in the cholesteryl ester transfer protein gene with angiographically assessed coronary atherosclerosis. Clin Genet. 2001; 60(5):374-380.
PUBMED
40. Volcik K, Ballantyne CM, Pownall HJ, Sharrett AR, Boerwinkle E. Interaction effects of high-density lipoprotein metabolism gene variation and alcohol consumption on coronary heart disease risk: the atherosclerosis risk in communities study. J Stud Alcohol Drugs. 2007;68(4):485-492.
PUBMED
41. Nettleton JA, Steffen LM, Ballantyne CM, Boerwinkle E, Folsom AR. Associations between HDL-cholesterol and polymorphisms in hepatic lipase and lipoprotein lipase genes are modified by dietary fat intake in African American and white adults. Atherosclerosis. 2007;194 (2):e131-e140.
PUBMED
42. Blankenberg S, Tiret L, Bickel C, et al. Genetic variation of the cholesterol ester transfer protein gene and the prevalence of coronary artery disease: the Athero-Gene case control study [in German]. Z Kardiol. 2004; 93(suppl 4):IV16-IV23.
PUBMED
43. Barzilai N, Atzmon G, Derby CA, Bauman JM, Lipton RB. A genotype of exceptional longevity is associated with preservation of cognitive function. Neurology. 2006; 67(12):2170-2175.
FREE FULL TEXT
44. Barzilai N, Atzmon G, Schechter C, et al. Unique lipoprotein phenotype and genotype associatedwith exceptional longevity. JAMA. 2003;290(15):2030- 2040.
FREE FULL TEXT
45. Bergman A, Atzmon G, Ye K, MacCarthy T, Barzilai N. Buffering mechanisms in aging: a systems approach toward uncovering the genetic component of aging. PLoS Comput Biol. 2007;3(8):e170.
PUBMED
46. Bauerfeind A, Knoblauch H, Schuster H, Luft FC, Reich JG. Single nucleotide polymorphism haplotypes in the cholesteryl-ester transfer protein (CETP) gene and lipid phenotypes. Hum Hered. 2002;54(4):166-173.
PUBMED
47. Bauerfeind A, Knoblauch H, Costanza MC, et al. Concordant association of lipid gene variation with a combined HDL/LDL-cholesterol phenotype in two European populations. Hum Hered. 2006;61(3):123- 131.
PUBMED
48. Bernstein MS, Costanza MC, James RW, et al. No physical activity x CETP 1b.-629 interaction effects on lipid profile. Med Sci Sports Exerc. 2003;35(7):1124-1129.
PUBMED
49. Klos KL, Sing CF, Boerwinkle E, et al. Consistent effects of genes involved in reverse cholesterol transport on plasma lipid and apolipoprotein levels in CARDIA participants. Arterioscler Thromb Vasc Biol. 2006; 26(8):1828-1836.
FREE FULL TEXT
50. Carr MC, Ayyobi AF, Murdoch SJ, Deeb SS, Brunzell JD. Contribution of hepatic lipase, lipoprotein lipase, and cholesteryl ester transfer protein to LDL andHDLheterogeneity in healthy women. Arterioscler Thromb Vasc Biol. 2002;22(4):667-673.
FREE FULL TEXT
51. Agerholm-Larsen B, Tybjaerg-Hansen A, Schnohr P, Steffensen R, Nordestgaard BG. Common cholesteryl ester transfer protein mutations, decreased HDL cholesterol, and possible decreased risk of ischemic heart disease: the Copenhagen City Heart Study. Circulation. 2000;102(18):2197-2203.
FREE FULL TEXT
52. Agerholm-Larsen B, Nordestgaard BG, Steffensen R, Jensen G, Tybjaerg-Hansen A. Elevated HDL cholesterol is a risk factor for ischemic heart disease in white womenwhen caused by a commonmutation in the cholesteryl ester transfer protein gene. Circulation. 2000; 101(16):1907-1912.
FREE FULL TEXT
53. Heidema AG, Feskens EJ, Doevendans PA, et al. Analysis of multiple SNPs in genetic association studies: comparison of three multi-locus methods to prioritize and select SNPs. Genet Epidemiol. 2007;31 (8):910-921.
PUBMED
54. Boer JM, Feskens EJ, Kuivenhoven JA, et al. Parental history of myocardial infarction: lipid traits, gene polymorphisms and lifestyle. Atherosclerosis. 2001;155 (1):149-156.
PUBMED
55. Chaaba R, Hammami S, Attia N, et al. Association of plasma cholesteryl ester transfer protein activity and polymorphism with coronary artery disease extent in Tunisian type II diabetic patients. Clin Biochem. 2005; 38(4):373-378.
PUBMED
56. Chen J, Yokoyama T, Saito K, et al. Association of human cholesteryl ester transfer protein-TaqI polymorphisms with serum HDL cholesterol levels in a normolipemic Japanese rural population. J Epidemiol. 2002; 12(2):77-84.
PUBMED
57. Choi HS, Park JB, Han KO, et al. A common mutation in cholesteryl ester transfer protein gene and plasma HDL cholesterol level before and after hormone replacement therapy in Korean postmenopausal women. Korean J Intern Med. 2002;17(2):83-87.
PUBMED
58. Corella D, Saiz C, Guillen M, et al. Association of TaqIB polymorphism in the cholesteryl ester transfer protein gene with plasma lipid levels in a healthy Spanish population. Atherosclerosis. 2000;152(2):367-376.
PUBMED
59. Ruiz-Narva ez EA, Yang Y, Nakanishi Y, Kirchdorfer J, Campos H. APOC3/A5 haplotypes, lipid levels, and risk of myocardial infarction in the Central Valley of Costa Rica. J Lipid Res. 2005;46(12):2605-2613.
FREE FULL TEXT
60. Cuchel M, Wolfe ML, deLemos AS, Rader DJ. The frequency of the cholesteryl ester transfer protein-TaqI B2 allele is lower in African Americans than in Caucasians. Atherosclerosis. 2002;163(1):169-174.
PUBMED
61. McCaskie PA, Beilby JP, Chapman CM, et al. Cholesteryl ester transfer protein gene haplotypes, plasma high-density lipoprotein levels and the risk of coronary heart disease. Hum Genet. 2007;121(3-4):401-411.
PUBMED
62. Gudnason V, Kakko S, Nicaud V, et al. Cholesteryl ester transfer protein gene effect on CETP activity and plasma high-density lipoprotein in European populations: the EARS Group. Eur J Clin Invest. 1999;29 (2):116-128.
PUBMED
63. Le Goff W, Guerin M, Nicaud V, et al. A novel cholesteryl ester transfer protein promoter polymorphism (-971G/A) associated with plasma high-density lipoprotein cholesterol levels: interaction with the TaqIB and -629C/A polymorphisms. Atherosclerosis. 2002; 161(2):269-279.
PUBMED
64. Corbex M, Poirier O, Fumeron F, et al. Extensive association analysis between the CETP gene and coronary heart disease phenotypes reveals several putative functional polymorphisms and gene-environment interaction. Genet Epidemiol. 2000;19(1):64-80.
PUBMED
65. Dachet C, Poirier O, Cambien F, Chapman J, Rouis M. New functional promoter polymorphism, CETP/-629, in cholesteryl ester transfer protein (CETP) gene related to CETP mass and high density lipoprotein cholesterol levels: role of Sp1/Sp3 in transcriptional regulation. Arterioscler Thromb Vasc Biol. 2000; 20(2):507-515.
FREE FULL TEXT
66. Fumeron F, Betoulle D, Luc G, et al. Alcohol intake modulates the effect of a polymorphism of the cholesteryl ester transfer protein gene on plasma high density lipoprotein and the risk of myocardial infarction. J Clin Invest. 1995;96(3):1664-1671.
PUBMED
67. Elosua R, Cupples LA, Fox CS, et al. Association between well-characterized lipoprotein-related genetic variants and carotid intimal medial thickness and stenosis: the Framingham Heart Study. Atherosclerosis. 2006; 189(1):222-228.
PUBMED
68. Ordovas JM, Cupples LA, Corella D, et al. Association of cholesteryl ester transfer protein-TaqIB polymorphism with variations in lipoprotein subclasses and coronary heart disease risk: the Framingham study. Arterioscler Thromb Vasc Biol. 2000;20(5):1323-1329.
FREE FULL TEXT
69. Freeman DJ, Griffin BA, Holmes AP, et al. Regulation of plasma HDL cholesterol and subfraction distribution by genetic and environmental factors: associations between the TaqI B RFLP in the CETP gene and smoking and obesity. Arterioscler Thromb. 1994; 14(3):336-344.
FREE FULL TEXT
70. Friedlander Y, Leitersdorf E, Vecsler R, Funke H, Kark J. The contribution of candidate genes to the response of plasma lipids and lipoproteins to dietary challenge. Atherosclerosis. 2000;152(1):239-248.
PUBMED
71. Goto A, Sasai K, Suzuki S, et al. Cholesteryl ester transfer protein and atherosclerosis in Japanese subjects: a study based on coronary angiography. Atherosclerosis. 2001;159(1):153-163.
PUBMED
72. Gudnason V, Thormar K, Humphries SE. Interaction of the cholesteryl ester transfer protein I405V polymorphism with alcohol consumption in smoking and non-smoking healthy men, and the effect on plasmaHDL cholesterol and apoAI concentration. Clin Genet. 1997; 51(1):15-21.
PUBMED
73. Hall WL, Vafeiadou K, Hallund J, et al. Soyisoflavone-enriched foods and markers of lipid and glu- cose metabolism in postmenopausal women: interactions with genotype and equol production. Am J Clin Nutr. 2006;83(3):592-600.
FREE FULL TEXT
74. Hall WL, Vafeiadou K, Hallund J, et al. Soyisoflavone-enriched foods and inflammatory biomarkers of cardiovascular disease risk in postmenopausal women: interactions with genotype and equol production. Am J Clin Nutr. 2005;82(6):1260-1268.
FREE FULL TEXT
75. Hannuksela ML, Liinamaa MJ, Kesaniemi YA, Savolainen MJ. Relation of polymorphisms in the cholesteryl ester transfer protein gene to transfer protein activity and plasma lipoprotein levels in alcohol drinkers. Atherosclerosis. 1994;110(1):35-44.
PUBMED
76. Lu H, Inazu A, Moriyama Y, et al. Haplotype analyses of cholesteryl ester transfer protein gene promoter: a clue to an unsolved mystery of TaqIB polymorphism. J Mol Med. 2003;81(4):246-255.
PUBMED
77. Hong SH, Kim YR, Song J, Kim JQ. Genetic variations of cholesterol ester transfer protein gene in Koreans. Hum Biol. 2001;73(6):815-821.
PUBMED
78. Bruce C, Sharp DS, Tall AR. Relationship of HDL and coronary heart disease to a common amino acid polymorphism in the cholesteryl ester transfer protein in men with and without hypertriglyceridemia. J Lipid Res. 1998;39(5):1071-1078.
FREE FULL TEXT
79. Curb JD, Abbott RD, Rodriguez BL, et al. A prospective study of HDL-C and cholesteryl ester transfer protein gene mutations and the risk of coronary heart disease in the elderly. J Lipid Res. 2004;45(5):948-953.
FREE FULL TEXT
80. Zhong S, Sharp DS, Grove JS, et al. Increased coronary heart disease in Japanese-American men with mutation in the cholesteryl ester transfer protein gene despite increased HDL levels. J Clin Invest. 1996;97 (12):2917-2923.
PUBMED
81. Pai JK, Pischon T, Ma J, et al. Inflammatory markers and the risk of coronary heart disease in men and women. N Engl J Med. 2004;351(25):2599-2610.
PUBMED
82. Hsu LA, Ko YL, Hsu KH, Ko YH, Lee YS. Genetic variations in the cholesteryl ester transfer protein gene and high density lipoprotein cholesterol levels in Taiwanese Chinese. Hum Genet. 2002;110(1):57-63.
PUBMED
83. Huang G-Y, Lu F-E, Li M-Z, et al. A comparative study on the allele frequencies of genetic variant Ile405Val of cholesteryl ester transfer protein and its influence on serum lipids in European and Asian populations. China J Modern Med. 1999;9(9): 9-12.
84. Hui SP. Frequency and effect on plasma lipoprotein metabolism of a mutation in the cholesteryl ester transfer protein gene in the Chinese [in Japanese]. Hokkaido Igaku Zasshi. 1997;72(3):319-327.
PUBMED
85. Horne BD, Camp NJ, Anderson JL, et al. Multiple less common genetic variants explain the association of the cholesteryl ester transfer protein gene with coronary artery disease. J Am Coll Cardiol. 2007;49(20): 2053-2060.
FREE FULL TEXT
86. Horne BD, Carlquist JF, Cannon-Albright LA, et al. High-resolution characterization of linkage disequilibrium structure and selection of tagging single nucleotide polymorphisms: application to the cholesteryl ester transfer protein gene. Ann Hum Genet. 2006; 70(pt 4):524-534.
PUBMED
87. Whiting BM, Anderson JL, Muhlestein JB, et al. Candidate gene susceptibility variants predict intermediate end points but not angiographic coronary artery disease. Am Heart J. 2005;150(2):243-250.
PUBMED
88. Ikewaki K, Mabuchi H, Teramoto T, et al. Association of cholesteryl ester transfer protein activity and TaqIB polymorphism with lipoprotein variations in Japanese subjects. Metabolism. 2003;52(12):1564-1570.
PUBMED
89. Inazu A, Nishimura Y, Terada Y, Mabuchi H. Effects of hepatic lipase gene promoter nucleotide variations on serumHDLcholesterol concentration in the general Japanese population. J Hum Genet. 2001;46 (4):172-177.
PUBMED
90. Keavney B, Palmer A, Parish S, et al. Lipid-related genes and myocardial infarction in 4685 cases and 3460 controls: discrepancies between genotype, blood lipid concentrations, and coronary disease risk. Int J Epidemiol. 2004;33(5):1002-1013.
FREE FULL TEXT
91. Juvonen T, Savolainen MJ, Kairaluoma MI, et al. Polymorphisms at the apoB, apoA-I, and cholesteryl ester transfer protein gene loci in patients with gallbladder disease. J Lipid Res. 1995;36(4):804-812.
ABSTRACT
92. Kaplan DB, Schreiber R, Oliveira HC, et al. Cholesteryl ester transfer protein gene mutations in Brazilian hyperalphalipoproteinemia. Clin Genet. 2006; 69(5):455-457.
PUBMED
93. Kark JD, Sinnreich R, Leitersdorf E, et al. Taq1B CETP polymorphism, plasma CETP, lipoproteins, apolipoproteins and sex differences in a Jewish population sample characterized by low HDL-cholesterol. Atherosclerosis. 2000;151(2):509-518.
PUBMED
94. Zheng KQ, Zhang SZ, He Y, et al. Association between cholesteryl ester transfer protein gene polymorphisms and variations in lipid levels in patients with coronary heart disease. Chin Med J (Engl). 2004;117 (9):1288-1292.
PUBMED
95. Kondo I, Berg K, Drayna D, Lawn R. DNA polymorphism at the locus for human cholesteryl ester transfer protein (CETP) is associated with high density lipoprotein cholesterol and apolipoprotein levels. Clin Genet. 1989;35(1):49-56.
PUBMED
96. Kuivenhoven JA, De KP, Boer JM, et al. Heterogeneity at the CETP gene locus: influence on plasma CETP concentrations and HDL cholesterol levels. Arterioscler Thromb Vasc Biol. 1997;17(3):560-568.
FREE FULL TEXT
97. Liu J, Zhao D, Liu S, et al. Study on the distribution and association of cholesteryl ester transfer protein-TaqIB polymorphism and plasma concentration in general population [in Chinese]. Zhonghua Liu Xing Bing Xue Za Zhi. 2003;24(4):300-303.
PUBMED
98. Lu F-E, Huang G-Y, Wiebusch H, Funke H, Assmann G. Allele frequencies of mutations 373 Ala - T polymorphism and coronary heart disease: does totality of evidence support causal role for homocysteine and preventive potential of folate? BMJ. 2005;331(7524):1053.
FREE FULL TEXT
24. Wheeler JG, Keavney BD, Watkins H, Collins R, Danesh J. Four paraoxonase gene polymorphisms in 11212 cases of coronary heart disease and 12786 controls: meta-analysis of 43 studies. Lancet. 2004; 363(9410):689-695.
PUBMED
25. Ye Z, Liu EH, Higgins JP, et al. Seven haemostatic gene polymorphisms in coronary disease: metaanalysis of 66,155 cases and 91,307 controls. Lancet. 2006;367(9511):651-658.
PUBMED
26. Samani NJ, Erdmann J, Hall AS, et al. Genomewide association analysis of coronary artery disease. N Engl J Med. 2007;357(5):443-453.
PUBMED
27. Wellcome Trust Case Control Consortium. Genomewide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 2007; 447(7145):661-678.
PUBMED
28. Higgins JP, Thompson SG, Deeks JJ, Altman DG. Measuring inconsistency in meta-analyses. BMJ. 2003; 327(7414):557-560.
FREE FULL TEXT
29. Egger M, Davey Smith G, Schneider M, Minder C. Bias in meta-analysis detected by a simple, graphical test. BMJ. 1997;315(7109):629-634.
FREE FULL TEXT
30. Thompson JF, Wood LS, Pickering EH, Dechairo B, Hyde CL. High-density genotyping and functional SNP localization in the CETP gene. J Lipid Res. 2007; 48(2):434-443.
FREE FULL TEXT
31. Thompson JF, Durham LK, Lira ME, Shear C, Milos PM. CETP polymorphisms associated with HDL cholesterol may differ from those associated with cardiovascular disease. Atherosclerosis. 2005;181(1): 45-53.
PUBMED
32. Lloyd DB, Lira ME, Wood LS, et al. Cholesteryl ester transfer protein variants have differential stability but uniform inhibition by torcetrapib. J Biol Chem. 2005; 280(15):14918-14922.
FREE FULL TEXT
33. Hinds DA, Seymour AB, Durham LK, et al. Application of pooled genotyping to scan candidate regions for association with HDL cholesterol levels. Hum Genomics. 2004;1(6):421-434.
PUBMED
34. Lira ME, Lloyd DB, Hallowell S, Milos PM, Thompson JF. Highly polymorphic repeat region in the CETP promoter induces unusual DNA structure. Biochim Biophys Acta. 2004;1684(1-3):38-45.
PUBMED
35. Thompson JF, Lloyd DB, Lira ME, Milos PM. Cholesteryl ester transfer protein promoter singlenucleotide polymorphisms in Sp1-binding sites affect transcription and are associatedwith high-density lipoprotein cholesterol. Clin Genet. 2004;66(3):223-228.
PUBMED
36. Akita H, Chiba H, Tsuchihashi K, et al. Cholesteryl ester transfer protein gene: two commonmutations and their effect on plasma high-density lipoprotein cholesterol content. J Clin Endocrinol Metab. 1994;79 (6):1615-1618.
ABSTRACT
37. Al-Daghri NM, Al-Attas O, Patel A, et al. Association between the cholesteryl ester transfer protein TaqIdetectable B polymorphism and low high-density lipoprotein cholesterol concentration in Saudis. Clin Sci (Lond). 2003;105(4):467-472.
PUBMED
38. Arai Y, Hirose N, Yamamura K, et al. Deficiency of choresteryl ester transfer protein and gene polymorphisms of lipoprotein lipase and hepatic lipase are not associated with longevity. J Mol Med. 2003;81(2): 102-109.
PUBMED
39. Arca M, Montali A, Ombres D, et al. Lack of association of the common TaqIB polymorphism in the cholesteryl ester transfer protein gene with angiographically assessed coronary atherosclerosis. Clin Genet. 2001; 60(5):374-380.
PUBMED
40. Volcik K, Ballantyne CM, Pownall HJ, Sharrett AR, Boerwinkle E. Interaction effects of high-density lipoprotein metabolism gene variation and alcohol consumption on coronary heart disease risk: the atherosclerosis risk in communities study. J Stud Alcohol Drugs. 2007;68(4):485-492.
PUBMED
41. Nettleton JA, Steffen LM, Ballantyne CM, Boerwinkle E, Folsom AR. Associations between HDL-cholesterol and polymorphisms in hepatic lipase and lipoprotein lipase genes are modified by dietary fat intake in African American and white adults. Atherosclerosis. 2007;194 (2):e131-e140.
PUBMED
42. Blankenberg S, Tiret L, Bickel C, et al. Genetic variation of the cholesterol ester transfer protein gene and the prevalence of coronary artery disease: the Athero-Gene case control study [in German]. Z Kardiol. 2004; 93(suppl 4):IV16-IV23.
PUBMED
43. Barzilai N, Atzmon G, Derby CA, Bauman JM, Lipton RB. A genotype of exceptional longevity is associated with preservation of cognitive function. Neurology. 2006; 67(12):2170-2175.
FREE FULL TEXT
44. Barzilai N, Atzmon G, Schechter C, et al. Unique lipoprotein phenotype and genotype associatedwith exceptional longevity. JAMA. 2003;290(15):2030- 2040.
FREE FULL TEXT
45. Bergman A, Atzmon G, Ye K, MacCarthy T, Barzilai N. Buffering mechanisms in aging: a systems approach toward uncovering the genetic component of aging. PLoS Comput Biol. 2007;3(8):e170.
PUBMED
46. Bauerfeind A, Knoblauch H, Schuster H, Luft FC, Reich JG. Single nucleotide polymorphism haplotypes in the cholesteryl-ester transfer protein (CETP) gene and lipid phenotypes. Hum Hered. 2002;54(4):166-173.
PUBMED
47. Bauerfeind A, Knoblauch H, Costanza MC, et al. Concordant association of lipid gene variation with a combined HDL/LDL-cholesterol phenotype in two European populations. Hum Hered. 2006;61(3):123- 131.
PUBMED
48. Bernstein MS, Costanza MC, James RW, et al. No physical activity x CETP 1b.-629 interaction effects on lipid profile. Med Sci Sports Exerc. 2003;35(7):1124-1129.
PUBMED
49. Klos KL, Sing CF, Boerwinkle E, et al. Consistent effects of genes involved in reverse cholesterol transport on plasma lipid and apolipoprotein levels in CARDIA participants. Arterioscler Thromb Vasc Biol. 2006; 26(8):1828-1836.
FREE FULL TEXT
50. Carr MC, Ayyobi AF, Murdoch SJ, Deeb SS, Brunzell JD. Contribution of hepatic lipase, lipoprotein lipase, and cholesteryl ester transfer protein to LDL andHDLheterogeneity in healthy women. Arterioscler Thromb Vasc Biol. 2002;22(4):667-673.
FREE FULL TEXT
51. Agerholm-Larsen B, Tybjaerg-Hansen A, Schnohr P, Steffensen R, Nordestgaard BG. Common cholesteryl ester transfer protein mutations, decreased HDL cholesterol, and possible decreased risk of ischemic heart disease: the Copenhagen City Heart Study. Circulation. 2000;102(18):2197-2203.
FREE FULL TEXT
52. Agerholm-Larsen B, Nordestgaard BG, Steffensen R, Jensen G, Tybjaerg-Hansen A. Elevated HDL cholesterol is a risk factor for ischemic heart disease in white womenwhen caused by a commonmutation in the cholesteryl ester transfer protein gene. Circulation. 2000; 101(16):1907-1912.
FREE FULL TEXT
53. Heidema AG, Feskens EJ, Doevendans PA, et al. Analysis of multiple SNPs in genetic association studies: comparison of three multi-locus methods to prioritize and select SNPs. Genet Epidemiol. 2007;31 (8):910-921.
PUBMED
54. Boer JM, Feskens EJ, Kuivenhoven JA, et al. Parental history of myocardial infarction: lipid traits, gene polymorphisms and lifestyle. Atherosclerosis. 2001;155 (1):149-156.
PUBMED
55. Chaaba R, Hammami S, Attia N, et al. Association of plasma cholesteryl ester transfer protein activity and polymorphism with coronary artery disease extent in Tunisian type II diabetic patients. Clin Biochem. 2005; 38(4):373-378.
PUBMED
56. Chen J, Yokoyama T, Saito K, et al. Association of human cholesteryl ester transfer protein-TaqI polymorphisms with serum HDL cholesterol levels in a normolipemic Japanese rural population. J Epidemiol. 2002; 12(2):77-84.
PUBMED
57. Choi HS, Park JB, Han KO, et al. A common mutation in cholesteryl ester transfer protein gene and plasma HDL cholesterol level before and after hormone replacement therapy in Korean postmenopausal women. Korean J Intern Med. 2002;17(2):83-87.
PUBMED
58. Corella D, Saiz C, Guillen M, et al. Association of TaqIB polymorphism in the cholesteryl ester transfer protein gene with plasma lipid levels in a healthy Spanish population. Atherosclerosis. 2000;152(2):367-376.
PUBMED
59. Ruiz-Narva ez EA, Yang Y, Nakanishi Y, Kirchdorfer J, Campos H. APOC3/A5 haplotypes, lipid levels, and risk of myocardial infarction in the Central Valley of Costa Rica. J Lipid Res. 2005;46(12):2605-2613.
FREE FULL TEXT
60. Cuchel M, Wolfe ML, deLemos AS, Rader DJ. The frequency of the cholesteryl ester transfer protein-TaqI B2 allele is lower in African Americans than in Caucasians. Atherosclerosis. 2002;163(1):169-174.
PUBMED
61. McCaskie PA, Beilby JP, Chapman CM, et al. Cholesteryl ester transfer protein gene haplotypes, plasma high-density lipoprotein levels and the risk of coronary heart disease. Hum Genet. 2007;121(3-4):401-411.
PUBMED
62. Gudnason V, Kakko S, Nicaud V, et al. Cholesteryl ester transfer protein gene effect on CETP activity and plasma high-density lipoprotein in European populations: the EARS Group. Eur J Clin Invest. 1999;29 (2):116-128.
PUBMED
63. Le Goff W, Guerin M, Nicaud V, et al. A novel cholesteryl ester transfer protein promoter polymorphism (-971G/A) associated with plasma high-density lipoprotein cholesterol levels: interaction with the TaqIB and -629C/A polymorphisms. Atherosclerosis. 2002; 161(2):269-279.
PUBMED
64. Corbex M, Poirier O, Fumeron F, et al. Extensive association analysis between the CETP gene and coronary heart disease phenotypes reveals several putative functional polymorphisms and gene-environment interaction. Genet Epidemiol. 2000;19(1):64-80.
PUBMED
65. Dachet C, Poirier O, Cambien F, Chapman J, Rouis M. New functional promoter polymorphism, CETP/-629, in cholesteryl ester transfer protein (CETP) gene related to CETP mass and high density lipoprotein cholesterol levels: role of Sp1/Sp3 in transcriptional regulation. Arterioscler Thromb Vasc Biol. 2000; 20(2):507-515.
FREE FULL TEXT
66. Fumeron F, Betoulle D, Luc G, et al. Alcohol intake modulates the effect of a polymorphism of the cholesteryl ester transfer protein gene on plasma high density lipoprotein and the risk of myocardial infarction. J Clin Invest. 1995;96(3):1664-1671.
PUBMED
67. Elosua R, Cupples LA, Fox CS, et al. Association between well-characterized lipoprotein-related genetic variants and carotid intimal medial thickness and stenosis: the Framingham Heart Study. Atherosclerosis. 2006; 189(1):222-228.
PUBMED
68. Ordovas JM, Cupples LA, Corella D, et al. Association of cholesteryl ester transfer protein-TaqIB polymorphism with variations in lipoprotein subclasses and coronary heart disease risk: the Framingham study. Arterioscler Thromb Vasc Biol. 2000;20(5):1323-1329.
FREE FULL TEXT
69. Freeman DJ, Griffin BA, Holmes AP, et al. Regulation of plasma HDL cholesterol and subfraction distribution by genetic and environmental factors: associations between the TaqI B RFLP in the CETP gene and smoking and obesity. Arterioscler Thromb. 1994; 14(3):336-344.
FREE FULL TEXT
70. Friedlander Y, Leitersdorf E, Vecsler R, Funke H, Kark J. The contribution of candidate genes to the response of plasma lipids and lipoproteins to dietary challenge. Atherosclerosis. 2000;152(1):239-248.
PUBMED
71. Goto A, Sasai K, Suzuki S, et al. Cholesteryl ester transfer protein and atherosclerosis in Japanese subjects: a study based on coronary angiography. Atherosclerosis. 2001;159(1):153-163.
PUBMED
72. Gudnason V, Thormar K, Humphries SE. Interaction of the cholesteryl ester transfer protein I405V polymorphism with alcohol consumption in smoking and non-smoking healthy men, and the effect on plasmaHDL cholesterol and apoAI concentration. Clin Genet. 1997; 51(1):15-21.
PUBMED
73. Hall WL, Vafeiadou K, Hallund J, et al. Soyisoflavone-enriched foods and markers of lipid and glu- cose metabolism in postmenopausal women: interactions with genotype and equol production. Am J Clin Nutr. 2006;83(3):592-600.
FREE FULL TEXT
74. Hall WL, Vafeiadou K, Hallund J, et al. Soyisoflavone-enriched foods and inflammatory biomarkers of cardiovascular disease risk in postmenopausal women: interactions with genotype and equol production. Am J Clin Nutr. 2005;82(6):1260-1268.
FREE FULL TEXT
75. Hannuksela ML, Liinamaa MJ, Kesaniemi YA, Savolainen MJ. Relation of polymorphisms in the cholesteryl ester transfer protein gene to transfer protein activity and plasma lipoprotein levels in alcohol drinkers. Atherosclerosis. 1994;110(1):35-44.
PUBMED
76. Lu H, Inazu A, Moriyama Y, et al. Haplotype analyses of cholesteryl ester transfer protein gene promoter: a clue to an unsolved mystery of TaqIB polymorphism. J Mol Med. 2003;81(4):246-255.
PUBMED
77. Hong SH, Kim YR, Song J, Kim JQ. Genetic variations of cholesterol ester transfer protein gene in Koreans. Hum Biol. 2001;73(6):815-821.
PUBMED
78. Bruce C, Sharp DS, Tall AR. Relationship of HDL and coronary heart disease to a common amino acid polymorphism in the cholesteryl ester transfer protein in men with and without hypertriglyceridemia. J Lipid Res. 1998;39(5):1071-1078.
FREE FULL TEXT
79. Curb JD, Abbott RD, Rodriguez BL, et al. A prospective study of HDL-C and cholesteryl ester transfer protein gene mutations and the risk of coronary heart disease in the elderly. J Lipid Res. 2004;45(5):948-953.
FREE FULL TEXT
80. Zhong S, Sharp DS, Grove JS, et al. Increased coronary heart disease in Japanese-American men with mutation in the cholesteryl ester transfer protein gene despite increased HDL levels. J Clin Invest. 1996;97 (12):2917-2923.
PUBMED
81. Pai JK, Pischon T, Ma J, et al. Inflammatory markers and the risk of coronary heart disease in men and women. N Engl J Med. 2004;351(25):2599-2610.
PUBMED
82. Hsu LA, Ko YL, Hsu KH, Ko YH, Lee YS. Genetic variations in the cholesteryl ester transfer protein gene and high density lipoprotein cholesterol levels in Taiwanese Chinese. Hum Genet. 2002;110(1):57-63.
PUBMED
83. Huang G-Y, Lu F-E, Li M-Z, et al. A comparative study on the allele frequencies of genetic variant Ile405Val of cholesteryl ester transfer protein and its influence on serum lipids in European and Asian populations. China J Modern Med. 1999;9(9): 9-12.
84. Hui SP. Frequency and effect on plasma lipoprotein metabolism of a mutation in the cholesteryl ester transfer protein gene in the Chinese [in Japanese]. Hokkaido Igaku Zasshi. 1997;72(3):319-327.
PUBMED
85. Horne BD, Camp NJ, Anderson JL, et al. Multiple less common genetic variants explain the association of the cholesteryl ester transfer protein gene with coronary artery disease. J Am Coll Cardiol. 2007;49(20): 2053-2060.
FREE FULL TEXT
86. Horne BD, Carlquist JF, Cannon-Albright LA, et al. High-resolution characterization of linkage disequilibrium structure and selection of tagging single nucleotide polymorphisms: application to the cholesteryl ester transfer protein gene. Ann Hum Genet. 2006; 70(pt 4):524-534.
PUBMED
87. Whiting BM, Anderson JL, Muhlestein JB, et al. Candidate gene susceptibility variants predict intermediate end points but not angiographic coronary artery disease. Am Heart J. 2005;150(2):243-250.
PUBMED
88. Ikewaki K, Mabuchi H, Teramoto T, et al. Association of cholesteryl ester transfer protein activity and TaqIB polymorphism with lipoprotein variations in Japanese subjects. Metabolism. 2003;52(12):1564-1570.
PUBMED
89. Inazu A, Nishimura Y, Terada Y, Mabuchi H. Effects of hepatic lipase gene promoter nucleotide variations on serumHDLcholesterol concentration in the general Japanese population. J Hum Genet. 2001;46 (4):172-177.
PUBMED
90. Keavney B, Palmer A, Parish S, et al. Lipid-related genes and myocardial infarction in 4685 cases and 3460 controls: discrepancies between genotype, blood lipid concentrations, and coronary disease risk. Int J Epidemiol. 2004;33(5):1002-1013.
FREE FULL TEXT
91. Juvonen T, Savolainen MJ, Kairaluoma MI, et al. Polymorphisms at the apoB, apoA-I, and cholesteryl ester transfer protein gene loci in patients with gallbladder disease. J Lipid Res. 1995;36(4):804-812.
ABSTRACT
92. Kaplan DB, Schreiber R, Oliveira HC, et al. Cholesteryl ester transfer protein gene mutations in Brazilian hyperalphalipoproteinemia. Clin Genet. 2006; 69(5):455-457.
PUBMED
93. Kark JD, Sinnreich R, Leitersdorf E, et al. Taq1B CETP polymorphism, plasma CETP, lipoproteins, apolipoproteins and sex differences in a Jewish population sample characterized by low HDL-cholesterol. Atherosclerosis. 2000;151(2):509-518.
PUBMED
94. Zheng KQ, Zhang SZ, He Y, et al. Association between cholesteryl ester transfer protein gene polymorphisms and variations in lipid levels in patients with coronary heart disease. Chin Med J (Engl). 2004;117 (9):1288-1292.
PUBMED
95. Kondo I, Berg K, Drayna D, Lawn R. DNA polymorphism at the locus for human cholesteryl ester transfer protein (CETP) is associated with high density lipoprotein cholesterol and apolipoprotein levels. Clin Genet. 1989;35(1):49-56.
PUBMED
96. Kuivenhoven JA, De KP, Boer JM, et al. Heterogeneity at the CETP gene locus: influence on plasma CETP concentrations and HDL cholesterol levels. Arterioscler Thromb Vasc Biol. 1997;17(3):560-568.
FREE FULL TEXT
97. Liu J, Zhao D, Liu S, et al. Study on the distribution and association of cholesteryl ester transfer protein-TaqIB polymorphism and plasma concentration in general population [in Chinese]. Zhonghua Liu Xing Bing Xue Za Zhi. 2003;24(4):300-303.
PUBMED
98. Lu F-E, Huang G-Y, Wiebusch H, Funke H, Assmann G. Allele frequencies of mutations 373 Ala - Pro and 451 Arg - Pro and 451 Arg - Gln in cholesterol ester transfer protein gene and their influences on lipid metabolism. Chin J Arterioscler. 1999;7(3):208-211.
99. Mendis S, Shepherd J, Packard CJ, Gaffney D. Genetic variation in the cholesteryl ester transfer protein and apolipoprotein A-I genes and its relation to coronary heart disease in a Sri Lankan population. Atherosclerosis. 1990;83(1):21-27.
PUBMED
100. Miltiadous G. HatzivassiliouM,Liberopoulos E, et al. Gene polymorphisms affecting HDL-cholesterol levels in the normolipidemic population. Nutr Metab Cardiovasc Dis. 2005;15(3):219-224.
PUBMED
101. Mitchell RJ, Earl L, Williams J, Bisucci T, Gasiamis H. Polymorphisms of the gene coding for the cholesteryl ester transfer protein and plasma lipid levels in Italian and Greek migrants to Australia.HumBiol. 1994; 66(1):13-25.
102. Motohashi Y, Maruyama T, Murata M, et al. Role of genetic factors (CETP gene Taq I B polymorphism and Apo A-I gene Msp I polymorphism) in serum HDL-C levels in women. Nutr Metab Cardiovasc Dis. 2004; 14(1):6-14.
PUBMED
103. Mukherjee M, Shetty KR. Variations in highdensity lipoprotein cholesterol in relation to physical activity and Taq 1B polymorphism of the cholesteryl ester transfer protein gene. Clin Genet. 2004;65(5): 412-418.
PUBMED
104. Noone E, Roche HM, Black I, Tully AM, Gibney MJ. Effect of postprandial lipaemia and Taq 1B polymorphism of the cholesteryl ester transfer protein (CETP) gene on CETP mass, activity, associated lipoproteins and plasma lipids. Br J Nutr. 2000;84(2):203-209.
PUBMED
105. Talmud PJ, Hawe E, Robertson K, et al. Genetic and environmental determinants of plasma high density lipoprotein cholesterol and apolipoprotein AI concentrations in healthy middle-aged men. Ann Hum Genet. 2002;66(pt 2):111-124.
PUBMED
106. Okumura K, Matsui H, Kamiya H, et al. Differential effect of two common polymorphisms in the cholesteryl ester transfer protein gene on low-density lipoprotein particle size. Atherosclerosis. 2002;161(2): 425-431.
PUBMED
107. Kakko S, Tamminen M, Paivansalo M, et al. Variation at the cholesteryl ester transfer protein gene in relation to plasma high density lipoproteins cholesterol levels and carotid intima-media thickness. Eur J Clin Invest. 2001;31(7):593-602.
PUBMED
108. Kakko S, Tamminen M, Paivansalo M, et al. Cholesteryl ester transfer protein gene polymorphisms are associated with carotid atherosclerosis in men. Eur J Clin Invest. 2000;30(1):18-25.
PUBMED
109. Kakko S, Tamminen M, Kesaniemi YA, Savolainen MJ. R451Qmutation in the cholesteryl ester transfer protein (CETP) gene is associated with high plasma CETP activity. Atherosclerosis. 1998;136(2):233-240.
PUBMED
110. Tamminen M, Kakko S, Kesaniemi YA, Savolainen MJ. A polymorphic site in the 3 Gln in cholesterol ester transfer protein gene and their influences on lipid metabolism. Chin J Arterioscler. 1999;7(3):208-211.
99. Mendis S, Shepherd J, Packard CJ, Gaffney D. Genetic variation in the cholesteryl ester transfer protein and apolipoprotein A-I genes and its relation to coronary heart disease in a Sri Lankan population. Atherosclerosis. 1990;83(1):21-27.
PUBMED
100. Miltiadous G. HatzivassiliouM,Liberopoulos E, et al. Gene polymorphisms affecting HDL-cholesterol levels in the normolipidemic population. Nutr Metab Cardiovasc Dis. 2005;15(3):219-224.
PUBMED
101. Mitchell RJ, Earl L, Williams J, Bisucci T, Gasiamis H. Polymorphisms of the gene coding for the cholesteryl ester transfer protein and plasma lipid levels in Italian and Greek migrants to Australia.HumBiol. 1994; 66(1):13-25.
102. Motohashi Y, Maruyama T, Murata M, et al. Role of genetic factors (CETP gene Taq I B polymorphism and Apo A-I gene Msp I polymorphism) in serum HDL-C levels in women. Nutr Metab Cardiovasc Dis. 2004; 14(1):6-14.
PUBMED
103. Mukherjee M, Shetty KR. Variations in highdensity lipoprotein cholesterol in relation to physical activity and Taq 1B polymorphism of the cholesteryl ester transfer protein gene. Clin Genet. 2004;65(5): 412-418.
PUBMED
104. Noone E, Roche HM, Black I, Tully AM, Gibney MJ. Effect of postprandial lipaemia and Taq 1B polymorphism of the cholesteryl ester transfer protein (CETP) gene on CETP mass, activity, associated lipoproteins and plasma lipids. Br J Nutr. 2000;84(2):203-209.
PUBMED
105. Talmud PJ, Hawe E, Robertson K, et al. Genetic and environmental determinants of plasma high density lipoprotein cholesterol and apolipoprotein AI concentrations in healthy middle-aged men. Ann Hum Genet. 2002;66(pt 2):111-124.
PUBMED
106. Okumura K, Matsui H, Kamiya H, et al. Differential effect of two common polymorphisms in the cholesteryl ester transfer protein gene on low-density lipoprotein particle size. Atherosclerosis. 2002;161(2): 425-431.
PUBMED
107. Kakko S, Tamminen M, Paivansalo M, et al. Variation at the cholesteryl ester transfer protein gene in relation to plasma high density lipoproteins cholesterol levels and carotid intima-media thickness. Eur J Clin Invest. 2001;31(7):593-602.
PUBMED
108. Kakko S, Tamminen M, Paivansalo M, et al. Cholesteryl ester transfer protein gene polymorphisms are associated with carotid atherosclerosis in men. Eur J Clin Invest. 2000;30(1):18-25.
PUBMED
109. Kakko S, Tamminen M, Kesaniemi YA, Savolainen MJ. R451Qmutation in the cholesteryl ester transfer protein (CETP) gene is associated with high plasma CETP activity. Atherosclerosis. 1998;136(2):233-240.
PUBMED
110. Tamminen M, Kakko S, Kesaniemi YA, Savolainen MJ. A polymorphic site in the 3 untranslated region of the cholesteryl ester transfer protein (CETP) gene is associated with low CETP activity. Atherosclerosis. 1996; 124(2):237-247.
PUBMED
111. Kauma H, Savolainen MJ, Heikkila R, et al. Sex difference in the regulation of plasma high density lipoprotein cholesterol by genetic and environmental factors. Hum Genet. 1996;97(2):156-162.
PUBMED
112. Padmaja N, Ravindra KM, Soya SS, Adithan C. Common variants of cholesteryl ester transfer protein gene and their association with lipid parameters in healthy volunteers of Tamilian population. Clin Chim Acta. 2007; 375(1-2):140-146.
PUBMED
113. Zee RY, Cook NR, Cheng S, et al. Multi-locus candidate gene polymorphisms and risk of myocardial infarction: a population-based, prospective genetic analysis. J Thromb Haemost. 2006;4(2):341-348.
PUBMED
114. Liu S, Schmitz C, Stampfer MJ, et al. A prospective study of TaqIB polymorphism in the gene coding for cholesteryl ester transfer protein and risk of myocardial infarction in middle-aged men [published correction appears in Atherosclerosis. 2003;166(2):415]. Atherosclerosis. 2002;161(2):469-474.
PUBMED
115. Zee RY, Cook NR, Cheng S, et al. Polymorphism in the P-selectin and interleukin-4 genes as determinants of stroke: a population-based, prospective genetic analysis. Hum Mol Genet. 2004;13(4):389-396.
FREE FULL TEXT
116. Plat J, Mensink RP. Relationship of genetic variation in genes encoding apolipoprotein A-IV, scavenger receptor BI, HMG-CoA reductase, CETP and apolipoprotein E with cholesterol metabolism and the response to plant stanol ester consumption. Eur J Clin Invest. 2002;32(4):242-250.
PUBMED
117. Borggreve SE, Hillege HL, Wolffenbuttel BH, et al. An increased coronary risk is paradoxically associated with common cholesteryl ester transfer protein gene variations that relate to higher high-density lipoprotein cholesterol: a population-based study. J Clin Endocrinol Metab. 2006;91(9):3382-3388.
FREE FULL TEXT
118. Asselbergs FW, Moore JH, van den Berg MP, et al. Arole for CETP TaqIB polymorphism in determining susceptibility to atrial fibrillation: a nested case control study. BMC Med Genet. 2006;7:39.
PUBMED
119. Borggreve SE, Hillege HL, Wolffenbuttel BH, et al. The effect of cholesteryl ester transfer protein -629C- untranslated region of the cholesteryl ester transfer protein (CETP) gene is associated with low CETP activity. Atherosclerosis. 1996; 124(2):237-247.
PUBMED
111. Kauma H, Savolainen MJ, Heikkila R, et al. Sex difference in the regulation of plasma high density lipoprotein cholesterol by genetic and environmental factors. Hum Genet. 1996;97(2):156-162.
PUBMED
112. Padmaja N, Ravindra KM, Soya SS, Adithan C. Common variants of cholesteryl ester transfer protein gene and their association with lipid parameters in healthy volunteers of Tamilian population. Clin Chim Acta. 2007; 375(1-2):140-146.
PUBMED
113. Zee RY, Cook NR, Cheng S, et al. Multi-locus candidate gene polymorphisms and risk of myocardial infarction: a population-based, prospective genetic analysis. J Thromb Haemost. 2006;4(2):341-348.
PUBMED
114. Liu S, Schmitz C, Stampfer MJ, et al. A prospective study of TaqIB polymorphism in the gene coding for cholesteryl ester transfer protein and risk of myocardial infarction in middle-aged men [published correction appears in Atherosclerosis. 2003;166(2):415]. Atherosclerosis. 2002;161(2):469-474.
PUBMED
115. Zee RY, Cook NR, Cheng S, et al. Polymorphism in the P-selectin and interleukin-4 genes as determinants of stroke: a population-based, prospective genetic analysis. Hum Mol Genet. 2004;13(4):389-396.
FREE FULL TEXT
116. Plat J, Mensink RP. Relationship of genetic variation in genes encoding apolipoprotein A-IV, scavenger receptor BI, HMG-CoA reductase, CETP and apolipoprotein E with cholesterol metabolism and the response to plant stanol ester consumption. Eur J Clin Invest. 2002;32(4):242-250.
PUBMED
117. Borggreve SE, Hillege HL, Wolffenbuttel BH, et al. An increased coronary risk is paradoxically associated with common cholesteryl ester transfer protein gene variations that relate to higher high-density lipoprotein cholesterol: a population-based study. J Clin Endocrinol Metab. 2006;91(9):3382-3388.
FREE FULL TEXT
118. Asselbergs FW, Moore JH, van den Berg MP, et al. Arole for CETP TaqIB polymorphism in determining susceptibility to atrial fibrillation: a nested case control study. BMC Med Genet. 2006;7:39.
PUBMED
119. Borggreve SE, Hillege HL, Wolffenbuttel BH, et al. The effect of cholesteryl ester transfer protein -629C- A promoter polymorphism on high-density lipoprotein cholesterol is dependent on serum triglycerides. J Clin Endocrinol Metab. 2005;90(7):4198-4204.
FREE FULL TEXT
120. Dullaart RP, Borggreve SE, Hillege HL, Dallinga-Thie GM. The association of HDL cholesterol concentration with the -629C A promoter polymorphism on high-density lipoprotein cholesterol is dependent on serum triglycerides. J Clin Endocrinol Metab. 2005;90(7):4198-4204.
FREE FULL TEXT
120. Dullaart RP, Borggreve SE, Hillege HL, Dallinga-Thie GM. The association of HDL cholesterol concentration with the -629C A CETP promoter polymorphism is not fully explained by its relationship with plasma cholesteryl ester transfer. Scand J Clin Lab Invest. 2008; 68(2):99-105.
PUBMED
121. van Himbergen TM, van der Schouw YT, Voorbij HA, et al. Paraoxonase (PON1) and the risk for coronary heart disease and myocardial infarction in a general population of Dutch women [published ahead of print December 26, 2007]. Atherosclerosis. 2007.122. Qin Q, Zhao B-R, Geng J, et al. The association of the lipoprotein lipase S447X and cholesteryl ester transfer protein TaqIB polymorphism with coronary heart disease. Chin J Cardiol. 2004;32(6):522-525.
123. Eiriksdottir G, Bolla MK, Thorsson B, et al. The -629C A CETP promoter polymorphism is not fully explained by its relationship with plasma cholesteryl ester transfer. Scand J Clin Lab Invest. 2008; 68(2):99-105.
PUBMED
121. van Himbergen TM, van der Schouw YT, Voorbij HA, et al. Paraoxonase (PON1) and the risk for coronary heart disease and myocardial infarction in a general population of Dutch women [published ahead of print December 26, 2007]. Atherosclerosis. 2007.122. Qin Q, Zhao B-R, Geng J, et al. The association of the lipoprotein lipase S447X and cholesteryl ester transfer protein TaqIB polymorphism with coronary heart disease. Chin J Cardiol. 2004;32(6):522-525.
123. Eiriksdottir G, Bolla MK, Thorsson B, et al. The -629C A polymorphism in the CETP gene does not explain the association of TaqIB polymorphism with risk and age of myocardial infarction in Icelandic men. Atherosclerosis. 2001;159(1):187-192.
PUBMED
124. Riemens SC, van Tol A, Stulp BK, Dullaart RP. Influence of insulin sensitivity and the TaqIB cholesteryl ester transfer protein gene polymorphism on plasma lecithin: cholesterol acyltransferase and lipid transfer protein activities and their response to hyperinsulinemia in non-diabetic men. J Lipid Res. 1999;40(8):1467-1474.
FREE FULL TEXT
125. Sandhofer A, Tatarczyk T, Laimer M, et al. The Taq1B-variant in the cholesteryl ester-transfer protein gene and the risk of metabolic syndrome [published ahead of print January 24, 2008]. Obesity (Silver Spring). 2008;16(4):919-922.
PUBMED
126. Weitgasser R, Galvan G, Malaimare L, et al. Cholesteryl ester transfer protein TaqIB polymorphism and its relation to parameters of the insulin resistance syndrome in an Austrian cohort. Biomed Pharmacother. 2004;58(10):619-627.
PUBMED
127. Arai H, Yamamoto A, Matsuzawa Y, et al. Polymorphisms in four genes related to triglyceride and HDL-cholesterol levels in the general Japanese population in 2000. J Atheroscler Thromb. 2005;12(5): 240-250.
PUBMED
128. Tai ES, Ordovas JM, Corella D, et al. The TaqIB and -629C A polymorphism in the CETP gene does not explain the association of TaqIB polymorphism with risk and age of myocardial infarction in Icelandic men. Atherosclerosis. 2001;159(1):187-192.
PUBMED
124. Riemens SC, van Tol A, Stulp BK, Dullaart RP. Influence of insulin sensitivity and the TaqIB cholesteryl ester transfer protein gene polymorphism on plasma lecithin: cholesterol acyltransferase and lipid transfer protein activities and their response to hyperinsulinemia in non-diabetic men. J Lipid Res. 1999;40(8):1467-1474.
FREE FULL TEXT
125. Sandhofer A, Tatarczyk T, Laimer M, et al. The Taq1B-variant in the cholesteryl ester-transfer protein gene and the risk of metabolic syndrome [published ahead of print January 24, 2008]. Obesity (Silver Spring). 2008;16(4):919-922.
PUBMED
126. Weitgasser R, Galvan G, Malaimare L, et al. Cholesteryl ester transfer protein TaqIB polymorphism and its relation to parameters of the insulin resistance syndrome in an Austrian cohort. Biomed Pharmacother. 2004;58(10):619-627.
PUBMED
127. Arai H, Yamamoto A, Matsuzawa Y, et al. Polymorphisms in four genes related to triglyceride and HDL-cholesterol levels in the general Japanese population in 2000. J Atheroscler Thromb. 2005;12(5): 240-250.
PUBMED
128. Tai ES, Ordovas JM, Corella D, et al. The TaqIB and -629C A polymorphisms at the cholesteryl ester transfer protein locus: associations with lipid levels in a multiethnic population: the 1998 Singapore National Health Survey. Clin Genet. 2003;63(1):19-30.
PUBMED
129. Song GJ, Han GH, Chae JJ, et al. The effects of the cholesterol ester transfer protein gene and environmental factors on the plasma high density lipoprotein cholesterol levels in the Korean population. Mol Cells. 1997;7(5):615-619.
PUBMED
130. Sorlí JV, Corella D, Francés F, et al. The effect of the APOE polymorphism on HDL-C concentrations depends on the cholesterol ester transfer protein gene variation in a Southern European population. Clin Chim Acta. 2006;366(1-2):196-203.
PUBMED
131. Deguchi H, Pecheniuk NM, Elias DJ, Averell PM, Griffin JH. High-density lipoprotein deficiency and dyslipoproteinemia associated with venous thrombosis in men. Circulation. 2005;112(6):893-899.
FREE FULL TEXT
132. Pecheniuk NM, Deguchi H, Elias DJ, Xu X, Griffin JH. Cholesteryl ester transfer protein genotypes associatedwith venous thrombosis and dyslipoproteinemia in males. J Thromb Haemost. 2006;4(9):2080-2082.
PUBMED
133. Tenkanen H, Koshinen P, Kontula K, et al. Polymorphisms of the gene encoding cholesterol ester transfer protein and serum lipoprotein levels in subjects with and without coronary heart disease. Hum Genet. 1991; 87(5):574-578.
PUBMED
134. Thompson JF, Lira ME, Durham LK, et al. Polymorphisms in the CETP gene and associationwith CETP mass and HDL levels. Atherosclerosis. 2003;167 (2):195-204.
PUBMED
135. Tobin MD, Braund PS, Burton PR, et al. Genotypes and haplotypes predisposing to myocardial infarction: a multilocus case-control study. Eur Heart J. 2004;25(6):459-467.
FREE FULL TEXT
136. Tsujita Y, Nakamura Y, Zhang Q, et al. The association between high-density lipoprotein cholesterol level and cholesteryl ester transfer protein TaqIB gene polymorphism is influenced by alcohol drinking in a population-based sample. Atherosclerosis. 2007;191(1): 199-205.
PUBMED
137. Hodo A polymorphisms at the cholesteryl ester transfer protein locus: associations with lipid levels in a multiethnic population: the 1998 Singapore National Health Survey. Clin Genet. 2003;63(1):19-30.
PUBMED
129. Song GJ, Han GH, Chae JJ, et al. The effects of the cholesterol ester transfer protein gene and environmental factors on the plasma high density lipoprotein cholesterol levels in the Korean population. Mol Cells. 1997;7(5):615-619.
PUBMED
130. Sorlí JV, Corella D, Francés F, et al. The effect of the APOE polymorphism on HDL-C concentrations depends on the cholesterol ester transfer protein gene variation in a Southern European population. Clin Chim Acta. 2006;366(1-2):196-203.
PUBMED
131. Deguchi H, Pecheniuk NM, Elias DJ, Averell PM, Griffin JH. High-density lipoprotein deficiency and dyslipoproteinemia associated with venous thrombosis in men. Circulation. 2005;112(6):893-899.
FREE FULL TEXT
132. Pecheniuk NM, Deguchi H, Elias DJ, Xu X, Griffin JH. Cholesteryl ester transfer protein genotypes associatedwith venous thrombosis and dyslipoproteinemia in males. J Thromb Haemost. 2006;4(9):2080-2082.
PUBMED
133. Tenkanen H, Koshinen P, Kontula K, et al. Polymorphisms of the gene encoding cholesterol ester transfer protein and serum lipoprotein levels in subjects with and without coronary heart disease. Hum Genet. 1991; 87(5):574-578.
PUBMED
134. Thompson JF, Lira ME, Durham LK, et al. Polymorphisms in the CETP gene and associationwith CETP mass and HDL levels. Atherosclerosis. 2003;167 (2):195-204.
PUBMED
135. Tobin MD, Braund PS, Burton PR, et al. Genotypes and haplotypes predisposing to myocardial infarction: a multilocus case-control study. Eur Heart J. 2004;25(6):459-467.
FREE FULL TEXT
136. Tsujita Y, Nakamura Y, Zhang Q, et al. The association between high-density lipoprotein cholesterol level and cholesteryl ester transfer protein TaqIB gene polymorphism is influenced by alcohol drinking in a population-based sample. Atherosclerosis. 2007;191(1): 199-205.
PUBMED
137. Hodo lugil U, Williamson DW, Huang Y, Mahley RW. An interaction between the TaqIB polymorphism of cholesterol ester transfer protein and smoking is associated with changes in plasma high-density lipoprotein cholesterol levels in Turks. Clin Genet. 2005; 68(2):118-127.
PUBMED
138. Vergani C, Lucchi T, Caloni M, et al. I405V polymorphism of the cholesteryl ester transfer protein (CETP) gene in young and very old people. Arch Gerontol Geriatr. 2006;43(2):213-221.
PUBMED
139. Lucchi T, Arosio B, Caloni M, et al. I405V polymorphism of the cholesteryl ester transfer protein gene in young and very old individuals [in Italian]. Ann Ital Med Int. 2005;20(1):45-50.
PUBMED
140. Martinelli N, Trabetti E, Bassi A, et al. The -1131 T lugil U, Williamson DW, Huang Y, Mahley RW. An interaction between the TaqIB polymorphism of cholesterol ester transfer protein and smoking is associated with changes in plasma high-density lipoprotein cholesterol levels in Turks. Clin Genet. 2005; 68(2):118-127.
PUBMED
138. Vergani C, Lucchi T, Caloni M, et al. I405V polymorphism of the cholesteryl ester transfer protein (CETP) gene in young and very old people. Arch Gerontol Geriatr. 2006;43(2):213-221.
PUBMED
139. Lucchi T, Arosio B, Caloni M, et al. I405V polymorphism of the cholesteryl ester transfer protein gene in young and very old individuals [in Italian]. Ann Ital Med Int. 2005;20(1):45-50.
PUBMED
140. Martinelli N, Trabetti E, Bassi A, et al. The -1131 T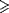 C and S19W APOA5 gene polymorphisms are associated with high levels of triglycerides and apolipoprotein C-III, but not with coronary artery disease: an angiographic study. Atherosclerosis. 2007;191(2): 409-417.
PUBMED
141. Vohl MC, Lamarche B, Pascot A, et al. Contribution of the cholesteryl ester transfer protein gene TaqIB polymorphism to the reduced plasma HDL-cholesterol levels found in abdominal obese menwith the features of the insulin resistance syndrome. Int J Obes Relat Metab Disord. 1999;23(9):918-925.
PUBMED
142. Wang W, Zhou X, Liu F, Hu H-N, Han D-F. Association of the TaqIB polymorphism and D442G mutation of cholesteryl ester transfer protein gene with coronary heart disease. Chin J Cardiol. 2004;32(11): 981-985.
143. Freeman DJ, Samani NJ, Wilson V, et al. A polymorphism of the cholesteryl ester transfer protein gene predicts cardiovascular events in non-smokers in the West of Scotland Coronary Prevention Study. Eur Heart J. 2003;24(20):1833-1842.
FREE FULL TEXT
144. WuY. Bai H, Liu R, Liu Y, Liu BW. Analysis of cholesterol ester transfer protein gene Taq IB and -629 C/A polymorphisms in patients with endogenous hypertriglyceridemia in Chinese population [in Chinese]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2006;23(6):640-646.
PUBMED
145. Yilmaz H, Isbir T, Agachan B, Karaali ZE. Effects of cholesterol ester transfer protein Taq1B gene polymorphism on serum lipoprotein levels in Turkish coronary artery disease patients. Cell Biochem Funct. 2005; 23(1):23-28.
PUBMED
146. Yilmaz H, Agachan B, Karaali ZE, Isbir T. Taq1B polymorphism of CETP gene on lipid abnormalities in patients with type II diabetes mellitus. Int J Mol Med. 2004;13(6):889-893.
PUBMED
147. Isbir T, Yilmaz H, Agachan B, Karaali ZE. Cholesterol ester transfer protein, apolipoprotein E and lipoprotein lipase genotypes in patients with coronary artery disease in the Turkish population. Clin Genet. 2003; 64(3):228-234.
PUBMED
148. Jang Y, Kim JY, Kim OY, et al. The -1131T C and S19W APOA5 gene polymorphisms are associated with high levels of triglycerides and apolipoprotein C-III, but not with coronary artery disease: an angiographic study. Atherosclerosis. 2007;191(2): 409-417.
PUBMED
141. Vohl MC, Lamarche B, Pascot A, et al. Contribution of the cholesteryl ester transfer protein gene TaqIB polymorphism to the reduced plasma HDL-cholesterol levels found in abdominal obese menwith the features of the insulin resistance syndrome. Int J Obes Relat Metab Disord. 1999;23(9):918-925.
PUBMED
142. Wang W, Zhou X, Liu F, Hu H-N, Han D-F. Association of the TaqIB polymorphism and D442G mutation of cholesteryl ester transfer protein gene with coronary heart disease. Chin J Cardiol. 2004;32(11): 981-985.
143. Freeman DJ, Samani NJ, Wilson V, et al. A polymorphism of the cholesteryl ester transfer protein gene predicts cardiovascular events in non-smokers in the West of Scotland Coronary Prevention Study. Eur Heart J. 2003;24(20):1833-1842.
FREE FULL TEXT
144. WuY. Bai H, Liu R, Liu Y, Liu BW. Analysis of cholesterol ester transfer protein gene Taq IB and -629 C/A polymorphisms in patients with endogenous hypertriglyceridemia in Chinese population [in Chinese]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2006;23(6):640-646.
PUBMED
145. Yilmaz H, Isbir T, Agachan B, Karaali ZE. Effects of cholesterol ester transfer protein Taq1B gene polymorphism on serum lipoprotein levels in Turkish coronary artery disease patients. Cell Biochem Funct. 2005; 23(1):23-28.
PUBMED
146. Yilmaz H, Agachan B, Karaali ZE, Isbir T. Taq1B polymorphism of CETP gene on lipid abnormalities in patients with type II diabetes mellitus. Int J Mol Med. 2004;13(6):889-893.
PUBMED
147. Isbir T, Yilmaz H, Agachan B, Karaali ZE. Cholesterol ester transfer protein, apolipoprotein E and lipoprotein lipase genotypes in patients with coronary artery disease in the Turkish population. Clin Genet. 2003; 64(3):228-234.
PUBMED
148. Jang Y, Kim JY, Kim OY, et al. The -1131T Cpolymorphism in the apolipoprotein A5 gene is associated with postprandial hypertriacylglycerolemia; elevated small, dense LDL concentrations; and oxidative stress in non obese Korean men. Am J Clin Nutr. 2004; 80(4):832-840.
FREE FULL TEXT
149. Dedoussis GV, Panagiotakos DB, Louizou E, et al. Cholesteryl ester-transfer protein (CETP) polymorphism and the association of acute coronary syndromes by obesity status in Greek subjects: the CARDIO2000-GENE study. Hum Hered. 2007;63(3-4): 155-161.
PUBMED
150. Falchi A, Piras IS, Vona G, et al. Cholesteryl ester transfer protein gene polymorphisms are associatedwith coronary artery disease in Corsican population (France). Exp Mol Pathol. 2007;83(1):25-29.
PUBMED
151. Falchi A, Giovannoni L, Piras IS, et al. Prevalence of genetic risk factors for coronary artery disease in Corsica island (France). Exp Mol Pathol. 2005;79(3): 210-213.
PUBMED
152. Andrikopoulos GK, Richter DJ, Needham EW, et al. Association of the ile405val mutation in cholesteryl ester transfer protein gene with risk of acute myocardial infarction. Heart. 2004;90(11):1336-1337.
FREE FULL TEXT
153. Morgan TM, Krumholz HM, Lifton RP, Spertus JA. Nonvalidation of reported genetic risk factors for acute coronary syndrome in a large-scale replication study. JAMA. 2007;297(14):1551-1561.
FREE FULL TEXT
154. van Acker BA, Botma GJ, Zwinderman AH, et al. High HDL cholesterol does not protect against coronary artery disease when associated with combined cholesteryl ester transfer protein and hepatic lipase gene variants [published ahead of print December 26, 2007]. Atherosclerosis. doi:10.1016/j.atherosclerosis.2007.11.019.155. Isaacs A, Sayed-Tabatabaei FA, Hofman A, et al. The cholesteryl ester transfer protein I405V polymorphism is associated with increased high-density lipoprotein levels and decreased risk of myocardial infarction: the Rotterdam Study. Eur J Cardiovasc Prev Rehabil. 2007;14(3):419-421.
FREE FULL TEXT
156. Isaacs A, Aulchenko YS, Hofman A, et al. Epistatic effect of cholesteryl ester transfer protein and hepatic lipase on serum high-density lipoprotein cholesterol levels. J Clin Endocrinol Metab. 2007;92(7): 2680-2687.
FREE FULL TEXT
157. Silva MC, Janssens AC, Hofman A, Witteman JC, Duijn CM. Cholesteryl ester transfer protein gene and coronary heart disease mortality: the Rotterdam Study. J Am Geriatr Soc. 2007;55(9):1483-1484.
PUBMED
158. Wu JH, Lee YT, Hsu HC, Hsieh LL. Influence of CETP gene variation on plasma lipid levels and coronary heart disease: a survey in Taiwan. Atherosclerosis. 2001;159(2):451-458.
PUBMED
159. Yamada Y, Izawa H, Ichihara S, et al. Prediction of the risk of myocardial infarction from polymorphisms in candidate genes. N Engl J Med. 2002; 347(24):1916-1923.
PUBMED
160. Yan S-K, Zhu Y-L, Cheng S, et al. Relationship between coronary heart disease and Taq IB &Msp I polymorphisms of cholesteryl ester transfer protein gene in Han nationality. Chin J Lab Med. 2004;27(10):671-675.
161. Zhang G-B, Jiang Z-W, Sun B-G, et al. Relationship of Taq IB polymorphism in the cholesteryl ester transfer protein gene to coronary artery disease. Chin J Arterioscler. 2005;13(1):88-90.
162. Zhao S-P, Li H, Xiao Z-J, Nie S. The effect of cholesteryl ester transfer protein TaqIB gene polymorphism on lipoprotein level. Chin J Cardiol. 2004; 32:816-818.
163. Zheng K, Zhang S, Zhang L, et al. Carriers of three polymorphisms of cholesteryl ester transfer protein gene are at increased risk to coronary heart disease in a Chinese population. Int J Cardiol. 2005;103(3):259-265.
PUBMED
164. Zheng KQ, Zhang SZ, Zhang L, et al. A novel missense mutation (L296 Q) in cholesteryl ester transfer protein gene related to coronary heart disease. Acta Biochim Biophys Sin (Shanghai). 2004;36(1):33-36.
PUBMED
165. Zheng KQ, Zhang SZ, Zhang KL, et al. Study on the association of cholesteryl ester transfer protein gene mutations with the susceptibility to coronary atherosclerotic heart disease [in Chinese]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2003;20(1):23-26.
PUBMED
166. Pharmacogenetics and risk of cardiovascular disease (PARC) resequencing project. http://droog.gs .washington.edu/parc/. Accessed January 23, 2008.167. Cardon LR, Palmer LJ. Population stratification and spurious allelic association. Lancet. 2003;361(9357): 598-604.
PUBMED
168. Keavney B, Danesh J, Parish S, et al. Fibrinogen and coronary heart disease: test of causality by ‘Mendelian randomization.’ Int J Epidemiol. 2006;35 (4):935-943.
FREE FULL TEXT
169. Davey Smith G, Ebrahim S. What can mendelian randomisation tell us about modifiable behavioural and environmental exposures? BMJ. 2005;330(7499): 1076-1079.
FREE FULL TEXT
170. Boekholdt SM, Thompson JF. Natural genetic variation as a tool in understanding the role of CETP in lipid levels and disease. J Lipid Res. 2003;44(6):1080-1093.
FREE FULL TEXT
171. Krishna R, Anderson MS, Bergman AJ, et al. Effect of the cholesteryl ester transfer protein inhibitor, anacetrapib, on lipoproteins in patients with dyslipidaemia and on 24-h ambulatory blood pressure in healthy individuals: two double-blind, randomised placebocontrolled phase I studies. Lancet. 2007;370(9603): 1907-1914.
PUBMED Cpolymorphism in the apolipoprotein A5 gene is associated with postprandial hypertriacylglycerolemia; elevated small, dense LDL concentrations; and oxidative stress in non obese Korean men. Am J Clin Nutr. 2004; 80(4):832-840.
FREE FULL TEXT
149. Dedoussis GV, Panagiotakos DB, Louizou E, et al. Cholesteryl ester-transfer protein (CETP) polymorphism and the association of acute coronary syndromes by obesity status in Greek subjects: the CARDIO2000-GENE study. Hum Hered. 2007;63(3-4): 155-161.
PUBMED
150. Falchi A, Piras IS, Vona G, et al. Cholesteryl ester transfer protein gene polymorphisms are associatedwith coronary artery disease in Corsican population (France). Exp Mol Pathol. 2007;83(1):25-29.
PUBMED
151. Falchi A, Giovannoni L, Piras IS, et al. Prevalence of genetic risk factors for coronary artery disease in Corsica island (France). Exp Mol Pathol. 2005;79(3): 210-213.
PUBMED
152. Andrikopoulos GK, Richter DJ, Needham EW, et al. Association of the ile405val mutation in cholesteryl ester transfer protein gene with risk of acute myocardial infarction. Heart. 2004;90(11):1336-1337.
FREE FULL TEXT
153. Morgan TM, Krumholz HM, Lifton RP, Spertus JA. Nonvalidation of reported genetic risk factors for acute coronary syndrome in a large-scale replication study. JAMA. 2007;297(14):1551-1561.
FREE FULL TEXT
154. van Acker BA, Botma GJ, Zwinderman AH, et al. High HDL cholesterol does not protect against coronary artery disease when associated with combined cholesteryl ester transfer protein and hepatic lipase gene variants [published ahead of print December 26, 2007]. Atherosclerosis. doi:10.1016/j.atherosclerosis.2007.11.019.155. Isaacs A, Sayed-Tabatabaei FA, Hofman A, et al. The cholesteryl ester transfer protein I405V polymorphism is associated with increased high-density lipoprotein levels and decreased risk of myocardial infarction: the Rotterdam Study. Eur J Cardiovasc Prev Rehabil. 2007;14(3):419-421.
FREE FULL TEXT
156. Isaacs A, Aulchenko YS, Hofman A, et al. Epistatic effect of cholesteryl ester transfer protein and hepatic lipase on serum high-density lipoprotein cholesterol levels. J Clin Endocrinol Metab. 2007;92(7): 2680-2687.
FREE FULL TEXT
157. Silva MC, Janssens AC, Hofman A, Witteman JC, Duijn CM. Cholesteryl ester transfer protein gene and coronary heart disease mortality: the Rotterdam Study. J Am Geriatr Soc. 2007;55(9):1483-1484.
PUBMED
158. Wu JH, Lee YT, Hsu HC, Hsieh LL. Influence of CETP gene variation on plasma lipid levels and coronary heart disease: a survey in Taiwan. Atherosclerosis. 2001;159(2):451-458.
PUBMED
159. Yamada Y, Izawa H, Ichihara S, et al. Prediction of the risk of myocardial infarction from polymorphisms in candidate genes. N Engl J Med. 2002; 347(24):1916-1923.
PUBMED
160. Yan S-K, Zhu Y-L, Cheng S, et al. Relationship between coronary heart disease and Taq IB &Msp I polymorphisms of cholesteryl ester transfer protein gene in Han nationality. Chin J Lab Med. 2004;27(10):671-675.
161. Zhang G-B, Jiang Z-W, Sun B-G, et al. Relationship of Taq IB polymorphism in the cholesteryl ester transfer protein gene to coronary artery disease. Chin J Arterioscler. 2005;13(1):88-90.
162. Zhao S-P, Li H, Xiao Z-J, Nie S. The effect of cholesteryl ester transfer protein TaqIB gene polymorphism on lipoprotein level. Chin J Cardiol. 2004; 32:816-818.
163. Zheng K, Zhang S, Zhang L, et al. Carriers of three polymorphisms of cholesteryl ester transfer protein gene are at increased risk to coronary heart disease in a Chinese population. Int J Cardiol. 2005;103(3):259-265.
PUBMED
164. Zheng KQ, Zhang SZ, Zhang L, et al. A novel missense mutation (L296 Q) in cholesteryl ester transfer protein gene related to coronary heart disease. Acta Biochim Biophys Sin (Shanghai). 2004;36(1):33-36.
PUBMED
165. Zheng KQ, Zhang SZ, Zhang KL, et al. Study on the association of cholesteryl ester transfer protein gene mutations with the susceptibility to coronary atherosclerotic heart disease [in Chinese]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2003;20(1):23-26.
PUBMED
166. Pharmacogenetics and risk of cardiovascular disease (PARC) resequencing project. http://droog.gs .washington.edu/parc/. Accessed January 23, 2008.167. Cardon LR, Palmer LJ. Population stratification and spurious allelic association. Lancet. 2003;361(9357): 598-604.
PUBMED
168. Keavney B, Danesh J, Parish S, et al. Fibrinogen and coronary heart disease: test of causality by ‘Mendelian randomization.’ Int J Epidemiol. 2006;35 (4):935-943.
FREE FULL TEXT
169. Davey Smith G, Ebrahim S. What can mendelian randomisation tell us about modifiable behavioural and environmental exposures? BMJ. 2005;330(7499): 1076-1079.
FREE FULL TEXT
170. Boekholdt SM, Thompson JF. Natural genetic variation as a tool in understanding the role of CETP in lipid levels and disease. J Lipid Res. 2003;44(6):1080-1093.
FREE FULL TEXT
171. Krishna R, Anderson MS, Bergman AJ, et al. Effect of the cholesteryl ester transfer protein inhibitor, anacetrapib, on lipoproteins in patients with dyslipidaemia and on 24-h ambulatory blood pressure in healthy individuals: two double-blind, randomised placebocontrolled phase I studies. Lancet. 2007;370(9603): 1907-1914.
PUBMED
ARTICLES EN RAPPORT
Cette semaine dans le JAMA Français
JAMA. 2008;299:2715.
Texte Complet
Gènes CETP, effets métaboliques et risque de maladie coronaire
Peter W. F. Wilson
JAMA. 2008;299:2795-2796.
Texte Complet
Polymorphismes du gène de la CETP : des nouvelles encourageantes
Philippe Gabriel Steg
JAMA. 2008;299:E1.
Texte Complet
|